Caracterização do gene mitocondrial citocromo b da espécie Odontesthes humensis
Vanessa Seidel1, Alexandra Möller Alves2, Gadriéli Cristina Gheno3, Rafael Aldrighi Tavares4, Mateus Tremea5, Suele Bueno dos Santos6, Suellen Susin Gazzola7, Sérgio Renato Noguez Piedras8
1 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
2 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
3 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
4 - Universidade Federal de Pelotas, UFPEL
5 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
6 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
7 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
8 - Universidade Federal de Pelotas, UFPEL
2 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
3 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
4 - Universidade Federal de Pelotas, UFPEL
5 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
6 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
7 - Universidade Federal de Santa Maria, UFSM/Campus Palmeira das Missões
8 - Universidade Federal de Pelotas, UFPEL
RESUMO -
Objetivou-se caracterizar o gene mitocondrial citocromo b da espécie Odontesthes humensis. A montagem por referência foi realizado com o programa Bowtie2, sendo utilizado como referência o gene mitocondrial citocromo b da espécie Odontesthes bonariensis. Os totais de 96 reads alinharam a 1104 pares de base do gene de referência, revelando 16 sítios de diferenciação entre as espécies. A partir do sequenciamento de nova geração caracterizou-se o gene mitocondrial citocromo b da espécie Odontesthes humensis, sendo de grande relevância para base de trabalhos futuros de conservação e melhoramento genético.
Palavras-chave: Sequenciamento, peixe-rei, CYTB.
Characterization of the Mitochondrial Cytochrome B gene of Odontesthes humensis specie
ABSTRACT - The aim of this study was try to characterize the Mitochondrial Cytochrome B gene of Odontesthes humensis. The assembly by reference was performed with the Bowtie2 program, using as reference the Mitochondrial Cytochrome B gene of the Odontesthes bonariensis species. The totals of 96 reads aligned to 1104 base pairs of the reference gene, revealing 16 spot of differentiation between species. From the sequencing of a new generation, we characterized the Mitochondrial Cytochrome B gene of Odontesthes humensis specie, being great relevance to the basis of future works in conservation and genetic improvement.Keywords: Sequencing, king fish, CYTB
Introdução
O peixe-rei possui pequeno porte, aproximadamente 30 centímetros na fase adulta, sua alimentação baseasse em moluscos, plâncton, microcrustáceos, algas e insetos pequenos. Possui habitat em estuários, mar aberto e local de água doce. No Brasil o peixe-rei encontra-se principalmente nas lagoas Mirim e Mangueira, sendo observada uma grande variedade de espécies como Odontesthes bonariensis, O. humensis, O. miriensis tendo como as O. bonariensis e O. humensis mais encontradas. (DYER, 2006). O cultivo da espécie de peixe-rei não é desenvolvido suficientemente para alcançar níveis comerciais, sendo sua comercialização através da coleta de populações naturais. O ponto inicial para que exista maior sucesso no cultivo da espécie é a consolidação de um programa de melhoramento genético. Para a implantação deste, deve-se ter como base uma grande variabilidade genética e a definição da espécie a ser produzida, alcançando-se assim as melhorias desejadas para gerações sucessivas. (TAVARES et al., 2011). É de grande importância entender e observar a caracterização de uma região mitocondrial, onde o sequenciamento nos mostra a grande variabilidade genética existente entre as espécies, podendo ser observadas evoluções cada vez mais detalhadamente. Baseados em DNA mitocondrial os marcadores moleculares possuem herança materna, contrariando os nucleares com herança biparental. A herança materna, por sua vez, esta conservada em muitos dos genes localizados no genoma mitocondrial, sendo relevante ao se considerar questões de filogenia e importância taxonômica. Um dos marcadores mitocondriais mais utilizados é o citocromo b (CYTB), enzima presente na cadeia respiratória mitocondrial e na cadeia respiratória do ciclo foto-redox, (MEYER, 1994). O objetivo do trabalho visa caracterizar o gene mitocondrial CYTB da espécie Odontesthes humensis, revelando a variabilidade existente no individuo, e consequentemente, na espécie.
Revisão Bibliográfica
O Brasil apresenta extensas áreas de reservatórios de água doce, equivalendo a 12% da água doce do planeta, clima favorável, terras disponíveis, crescente mercado interno e maior biodiversidade de espécies nativas de peixes, contudo sua produção aquícola é irrisória (IBAMA, 2008; RESENDE et al., 2008). Além disso, a tilápia (Oreochromis niloticus) é a espécie mais representativa (KUBITZA et al., 2012). Porém, é uma espécie exótica e através da piscicultura pode ocorrer à introdução desta no ambiente, mudando a ecologia local, competindo por espaço, alimento com as espécies nativas e introduzindo patógenos e parasitas. A forma para superar este problema é através do desenvolvimento da piscicultura de espécies nativas (SANDRE, 2009). O Rio Grande do Sul apresenta uma situação privilegiada, com reservatórios naturais e artificiais de água doce, além de extensas áreas de terras de várzeas e espécies nativas que merecem destaques, como o jundiá (Rhamdia quelen) e o peixe-rei (Odontesthes spp.). O peixe-rei é um representante da ordem atheriniformes que inclui peixes encontrados em águas doces, marinhas, tropicais e temperadas. Dentro dos atheriniformes é um exemplar da subfamília atherinopsinae, que são compostos por seis gêneros em dois grupos antitropicais: Atherinopsini na América do Norte (Atherinops, Atherinopsis, Colpichtys, Leuresthes) e Sorgentinini na América do Sul (Basilichthys, Odontesthes) (DYER, 2006). O gênero Odontesthes apresenta a maioria das espécies e mais amplamente distribuída, sendo encontradas em águas costeiras marinhas, lagoas de água doce (BEMVENUTI, 2006) e estocada com sucesso em bacias artificiais de países sul-americanos. A elevada qualidade do peixe-rei, boa aceitação comercial, carne com sabor, cheiro, textura e características químicas semelhantes às das requintadas espécies marinhas (SOMOZA et al., 2008), também tem incentivado o cultivo em locais distantes da sua área de distribuição nativa, como o Japão e a Itália (BAIGÚN et al., 2009). Na bacia da lagoa Mirim a espécie mais comum é O. bonariensis, vulgarmente conhecida como "bicudo", apresentando, de acordo com DE BUEN (1953), corpo alto, espesso e relativamente curto, com marcada curva ventral, cabeça longa com extremidade proeminente e ligeiramente convexa para cima, e boca excessivamente protáctil. Seu cultivo não é desenvolvido o suficiente para alcançar níveis comerciais, sendo a comercialização desta espécie através da coleta de populações naturais. O ponto inicial para o sucesso do cultivo de peixe-rei é a consolidação de um programa de melhoramento genético (TAVARES et al., 2011). Ao se realizar este tipo de técnica, com base em uma grande variabilidade genética inicial, asseguram-se a existência de variabilidade suficiente para se alcançar as melhorias desejadas para sucessivas gerações (KUBITZA et al., 2007). Marcadores moleculares baseados em DNA mitocondrial (mtDNA) possuem apenas herança materna, ao contrário dos nucleares que possuem herança biparental. De acordo com OLSON et al. (2009) por à herança materna estar altamente conservada em muitos dos genes localizados no genoma mitocondrial, estes marcadores, muitas vezes podem ser usados para resolver as relações que medem períodos de tempo muito longos e são relevantes quando se considera questões de filogenia e importância taxonômica. Segundo SILVA et al. (2012), nas últimas décadas as técnicas de biologia molecular vêm ganhando espaço nos estudos de identificação molecular e, com os seus últimos avanços é possível comparar sequências de nucleotídeos do DNA, principalmente em regiões codificadoras de genes específicos, considerada conservada entre as espécies em estudo (CHUEIRE et al., 2003), podendo usar esta informação nos estudos de identificação de espécies. Um dos marcadores moleculares bastante conhecido e utilizado é o citocromo b que é a subunidade catalítica central da ubiquinol citocromo c reductase, uma enzima que está presente na cadeia respiratória da mitocôndria e na cadeia respiratória do ciclo foto-redox de muitas bactérias (MEYER, 1994), todos os organismos eucariotos necessitam desta classe de enzima redox, e consequentemente do citocromo b para a conversão de energia (ORREGO, 2012). Seu uso se justifica pela presença de regiões conservadas e variáveis, as quais contem sinais que podem ser utilizados em análises filogenéticas em diferentes níveis taxonômicos, sendo considerado excelente marcador molecular, amplamente utilizado como ferramenta em filogenias moleculares (ORREGO, 2012).Materiais e Métodos
A biblioteca de DNA, da espécie Odontesthes Humensis foi obtida a partir de um sequenciador GAIIx (Illumina, USA) no modo paired-end, para a obtenção de sequências com 150 pares de bases (pb). A qualidade de cada read foi analisada com o programa FastQC (PATEL e JAIN, 2012). Após a análise de qualidade foi realizada a remoção dos adaptadores e a remoção de read de baixa qualidade com o programa Trimmomatic (BOLGER et al., 2014). As sequências das extremidades dos reads foram removidas quando as médias de qualidade fossem inferiores a Phread 15 em intervalos de quatro bases. Também foram removidos os reads com comprimentos menores que 32pb. Para a verificação da eficiência da filtragem foi utilizado novamente o programa FastQC. A montagem por referência foi realizado com o programa Bowtie2 (LANGMEAD e SALZBERG, 2012) sendo utilizado como referência o gene mitocondrial CYTB da espécie Odontesthes Bonariensis (GenBank: 400709.1) com comprimento de 1104pb. O arquivo no formato SAM, gerado na montagem, foi transformado em formato BAM com o programa Samtoll (LI et al., 2009) e posteriormente as reads montadas contra a referência foram visualizadas com o programa Tablet (MILNE et al., 2013).Resultados e Discussão
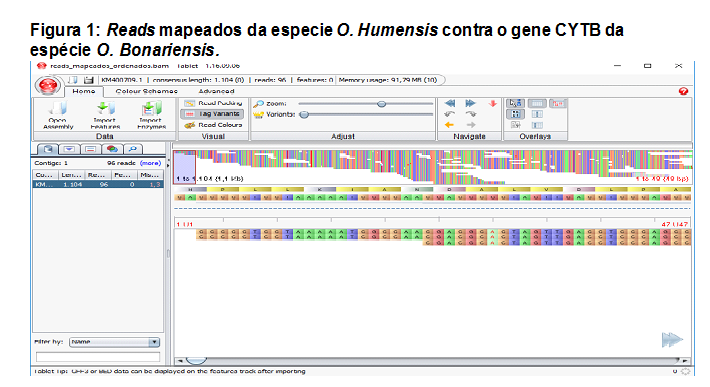
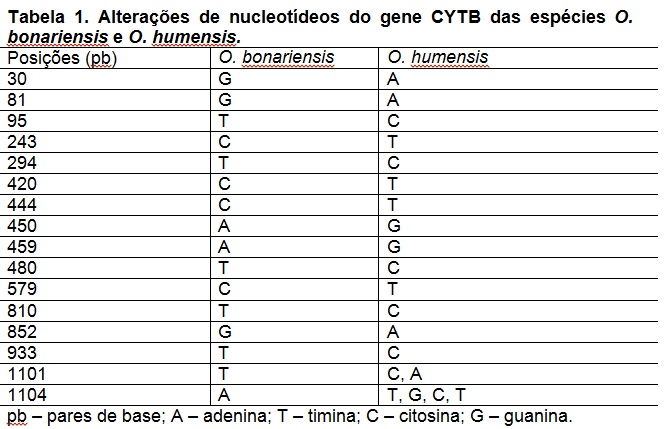
O sequenciamento do DNA resultou em aproximadamente 1.265.204 reads, sendo estes de 35 a 151pb, subdivididos em dois arquivos paired-end. Subsequentemente fez-se a filtragem através do programa Trimmomatic mantendo-se 1.227.534 reads e sendo removidos 37.670 reads. As bases que restaram apresentam-se de boa qualidade, com valores de Phred acima de 24. Segundo DEL FABBRO et al. (2013), ocorre o aumento das taxas de alinhamento dos reads a um genoma de referência, mesmo com a diminuição do volume de sequências após a filtragem. Os totais de 96 reads alinharam a 1.104 pares de base do gene de referência CYTB de O. bonariensis (figura 1), revelando 16 sítios de diferenciação entre as espécies (tabela 1). Segundo ORREGO (2012), todos os organismos eucariontes utilizam o CYTB para a conversão de energia, apresentando regiões variáveis e conservadas, utilizadas em análises filogenéticas com diferentes níveis taxonômicos.Conclusões
A partir do sequenciamento de nova geração identificou-se o gene mitocondrial citocromo b da espécie Odontesthes humensis a partir do gene mitocondrial citocromo b da espécie Odontesthes bonariensis. É de grande relevância caracterizar regiões mitocondriais para base de trabalhos futuros de conservação e melhoramento genético.Gráficos e Tabelas