IDENTIFICAÇÃO DO GENE MITOCONDRIAL CITOCROMO B DA ESPÉCIE PIRACANJUBA (Brycon orbignyanus)
Alexandra Moller Alves1, Gadriéli Cristina Gheno2, Heden Luiz Marques Moreira3, Mateus Tremea4, Rafael Aldrighi Tavares5, Suele Bueno dos Santos6, Suellen Susin Gazzola7, Vanessa Seidel8
1 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
2 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
3 - Universidade Federal de Pelotas/UFPEL
4 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
5 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
6 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
7 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
8 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
2 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
3 - Universidade Federal de Pelotas/UFPEL
4 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
5 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
6 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
7 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
8 - Universidade Federal de Santa Maria/UFSM/Campus Palmeira das Missões
RESUMO -
Objetivou-se identificar a região mitocondrial Citocromo b da espécie Brycon orbignyanus. O programa Bowtie2 foi utilizado para realizar a montagem de referência, o gene mitocondrial utilizado como referência foi o da espécie Brycon hilarii. Um total de 96 reads foram alinhados a 1104 pares de base do gene de referência, revelando 68 sítios de diferenciação entre as espécies. A partir do sequenciamento de nova geração foi identificado o gene mitocondrial Citocromo b da espécie Brycon orbignyanus, sendo de grande relevância para base de trabalhos futuros de conservação e melhoramento genético.
Palavras-chave: Palavras-chave: DNA mitocondrial; sequenciamento; Piracanjuba.
IDENTIFICATION OF THE MITOCHONDRIAL GENE CYTOCHROME B OF THE SPECIES PIRACANJUBA (Brycon orbignyanus)
ABSTRACT - Abstract: The objective of the study was to identify the Cytochrome b mitochondrial region on the Brycon orbignyanus. The Bowtie2 program was applied to do the reference assembly and the mitochondrial gene used as reference was the Brycon hilarii. A number of 96 reads was aligned to 1104 base pairs of the reference gene, revealing 68 differentiation sites between the species. From the sequencing of the new generation, the Cytochrome b mitochondrial gene on Brycon orbignyanus was identified, having great relevance as a basis for future works on conservation and genetic improvement.Keywords: Keywords: mitochondrial DNA; Sequencing; Piracanjuba.
Introdução
Tratando-se de uma espécie em risco de extinção, devido aos impactos causados em seu hábitat, a Piracanjuba, Brycon orbignyanus, é um peixe conhecido pelo seu tom avermelhado e de sabor semelhante ao Salmão. A espécie é encontrada em regiões do Mato Grosso do Sul, São Paulo, Minas Gerais, Paraná e Goiás, sendo evidente em águas claras, canais de rios, em áreas próximas às margens e locais de corredeiras (ABIMORAD, 2015). A mitocôndria é uma organela que, diferente das outras, possui sua própria carga genética (mtDNA). O DNA mitocondrial retrata apenas 1 a 2% do DNA celular, em duplo filamento circular, codificando apenas 37 genes. Os marcadores moleculares dispõe da herança materna, ou seja, são baseados em mtDNA (OLSON et al., 2009). O objetivo do trabalho visa identificar o citocomo b (cytb) da espécie Brycon orbignyanus, com grande relevância a caracterização do DNA mitocondrial, revelando a variabilidade existente no individuo, e consequentemente, na espécie.Revisão Bibliográfica
A piracanjuba, Brycon orbignyanus (Characiformes, Characidae, Bryconinae) também conhecida como bracanjuva, é uma espécie nativa das bacias formadas pelos rios Uruguai e Paraná (ZANIBONI-FILHO; REYNALTE-TATAJE; WEINGARTNER, 2006). Devido ao seu elevado valor comercial, qualidade da carne, precocidade, adaptação ao cativeiro e voracidade e briga na pesca esportiva é uma espécie de grande interesse por produtores e pescadores (BORBA; FRACALOSSI; PEZZATO, 2006; CECCARELLI; SENHORINI; RÊGO, 2005).Todavia, a diminuição de suas populações naturais em varias bacias hidrográficas – catalogada como espécie em risco de extinção (IBAMA, 2012) e a perda de variabilidade genética pode levar a depressão por endogamia que causa diminuição no desempenho e sobrevivência de progênies, incluindo aquelas liberadas em programas de conservação (PEREZ-ENRIQUEZ et al., 2009; POVH et al., 2008). Das intervenções realizadas para amenizar a diminuição de populações naturais de peixes destacam-se os programas de repovoamento (LOPERABARRERO; RIBEIRO; POVH, 2007). De acordo com Kalinowski et al. (2012), as principais metas que um programa de repovoamento deve alcançar quando se trata de peixes em risco de extinção, como é o caso do B. orbignyanus, são: 1) evitar a perda completa do grupo de genes; 2) conservar o máximo possível a diversidade genética; 3) realizar as metas 1 e 2 sem comprometer a sobrevivência das populações através do tempo. Assim, as introduções de peixes sucedidas de forma errônea e sem apoio científico pode resultar na redução da variabilidade genética nas populações naturais, devido principalmente a manejos reprodutivos realizados de forma errada nos estoques, os quais podem reduzir o potencial evolutivo e a resposta a modificações ambientais das progênies (POVH et al., 2011; RICHARDS; WARES; MACKIE, 2010). Desta forma, segue sendo necessário o programa de repovoamento e o estudo dos marcadores mitocôndrias. Por apresentar uma taxa de mutação quatro vezes maior, quando comparado com o DNA nuclear, o DNA mitocondrial torna-se popular em estudos de repovoamento (ROSA; PAIVA, 2009) Um dos marcadores mitocondriais mais utilizados é o citocromo b (CYTB), enzima presente na cadeia respiratória mitocondrial e na cadeia respiratória do ciclo foto-redox, (MEYER, 1994). O citocromo b é a subunidade catalítica central da ubiquinol citocromo c reductase, uma enzima que está presente na cadeia respiratória da mitocôndria e na cadeia respiratória do ciclo foto-redox de muitas bactérias (MEYER, 1994), todos os organismos eucariotos necessitam desta classe de enzima redox, e consequentemente do citocromo b para a conversão de energia (ORREGO, 2012). Seu uso se justifica pela presença de regiões conservadas e variáveis, as quais contem sinais que podem ser utilizados em análises filogenéticas em diferentes níveis taxonômicos, sendo considerado excelente marcador molecular, amplamente utilizado como ferramenta em filogenias moleculares (ORREGO, 2012).Materiais e Métodos
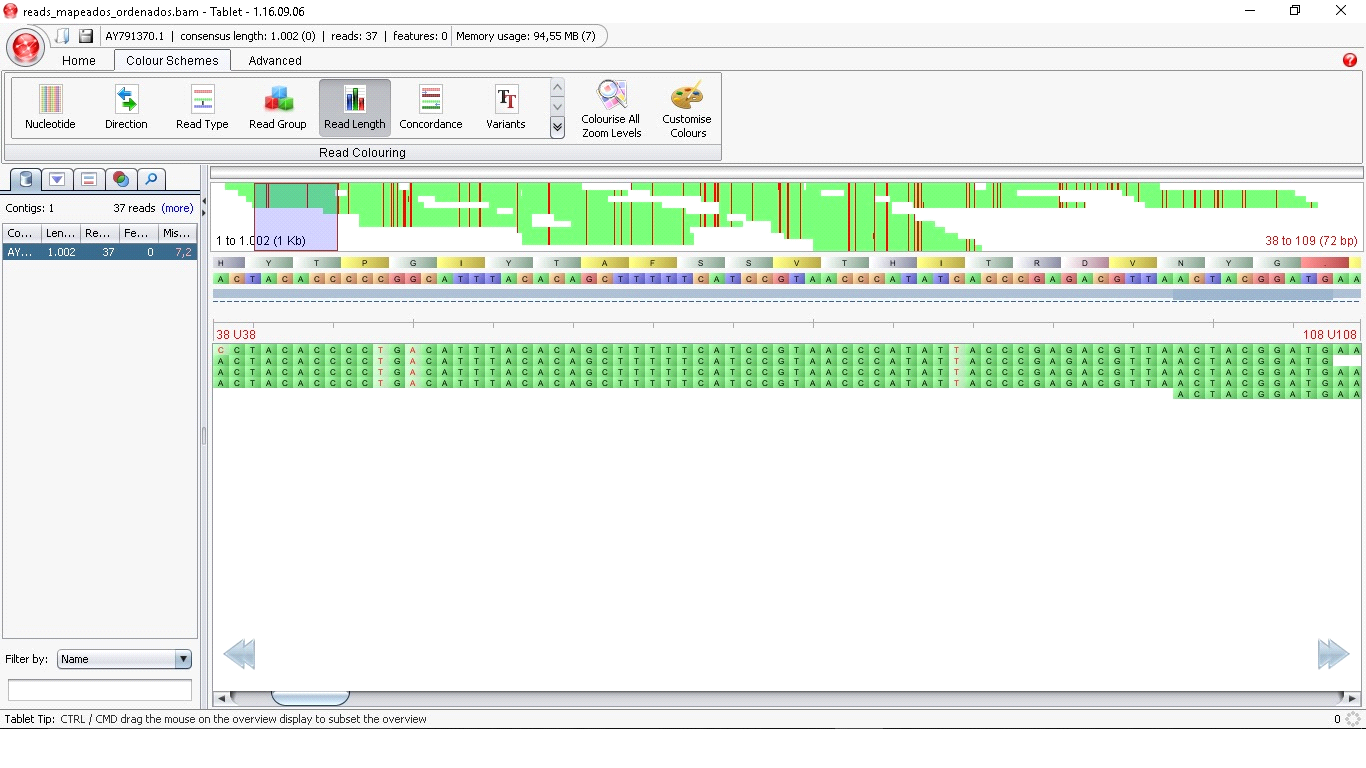
A biblioteca de DNA, da espécie Brycon orbignyanus foi obtida a partir de um sequenciador GAIIx (Illumina, USA) no modo paired-end, para a obtenção de sequências com 150 pares de bases (pb). A qualidade de cada read foi analisada com o programa FastQC (PATEL e JAIN, 2012). Após a análise de qualidade foi realizada a remoção dos adaptadores Illumina e a remoção de read de baixa qualidade com o programa Trimmomatic (BOLGER et al., 2014). As sequências das extremidades dos reads foram removidas quando as médias de qualidade fossem inferiores a Phread 15 em intervalos de quatro bases. Também foram removidos os reads com comprimentos menores que 32pb. Para a verificação da eficiência da filtragem foi utilizado novamente o programa FastQC. A montagem por referência foi realizado com o programa Bowtie2 (Langmead e Salzberg, 2012) sendo utilizado como referência o gene mitocondrial CYTB (GenBank: AY791370 ) com comprimento de 1104pb da espécie Brycon hilarii. O arquivo no formato SAM, gerado na montagem, foi transformado em formato BAM com o programa Samtoll (LI et al., 2009) e posteriormente as reads montadas contra a referência foram visualizadas com o programa Tablet (MILNE et al., 2013).Resultados e Discussão
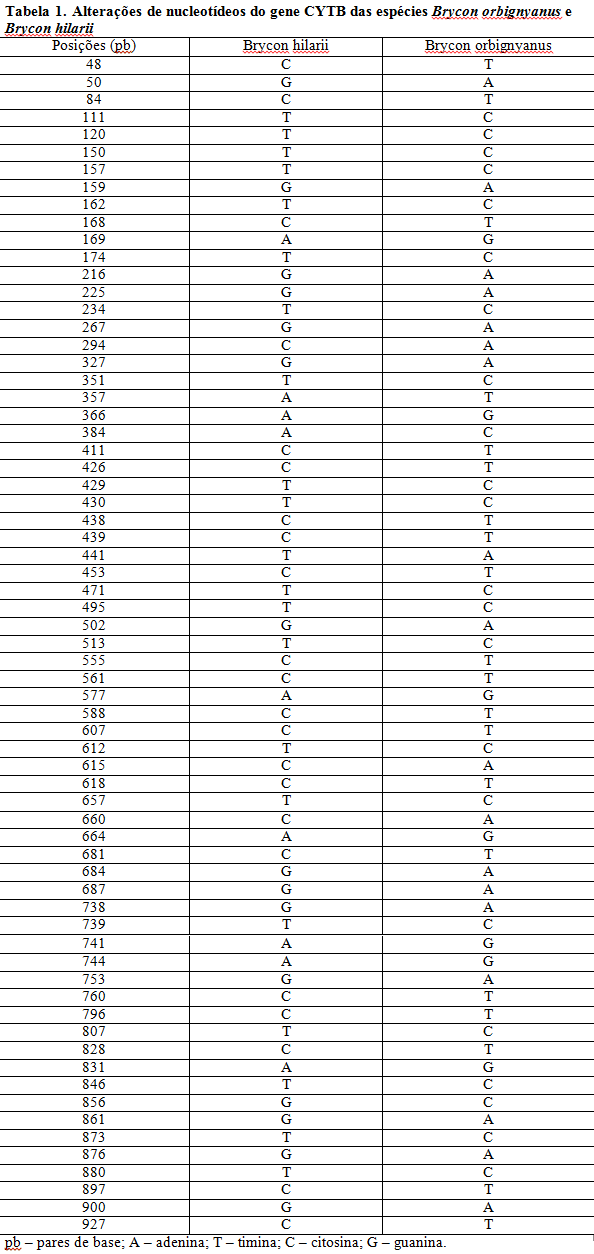
O sequenciamento do DNA resultou em torno de 820.390 reads, sendo estes de 35 a 151 pares de base, subdivididos em dois arquivos Paired-ends. Posteriormente realizou-se a filtragem através do programa Trimmomatic mantendo-se 794.399 reads sendo removidos 25.991, tendo-se os reads restantes valores de phred acima de 24. Após a montagem do número total de reads filtrados contra o gene de referência do CYTB de Brycon hilarii obteve-se 96 reads alinhados a 1104 pares de base (figura 1). Ao se realizar este tipo de técnica, com base em uma grande variabilidade genética inicial, asseguram-se a existência de variabilidade suficiente para se alcançar as melhorias desejadas para sucessivas gerações (KUBITZA et al., 2007). A análise da comparação do DNA mitocondrial das espécies Brycon hilarii e Brycon orbignyanus, gerou resultados com alta taxa de variação, sendo encontrados 68 sítios de variação. Espécies com maior variabilidade genética terão maiores chances de se recuperar tanto em número de indivíduos quanto com relação à resistência perante as adversidades do meio ambiente (PAES, 2013). Populações congênitas possuem altos níveis de variação genética. Essas variações são introduzidas nas espécies continuamente por mutação ou migração de indivíduos de outras populações. A variação genética em uma população pode ser medida pela heterozigosidade observada (ho), sendo esta referente a porcentagem de indivíduos heterozigotos em determinados loci gênicos. Portanto, a variabilidade genética é essencial para a permanência evolutiva das espécies.Conclusões
Com base no sequenciamento identificou-se o gene mitocondrial CYTB da espécie Brycon orbignyanus a partir do gene referente Brycon hilarii. É de grande significância identificar e descrever as regiões mitocondriais para base de trabalhos de conservação de populações naturais de peixes, aumentando a variabilidade e promovendo aprimoramentos estimados em gerações futuras da espécie.Gráficos e Tabelas