POLIMORFISMO DO GENE MITOCONDRIAL CITOCROMO B DA ESPÉCIE JURUPOCA (Hemisorubim phatyrhynchos).
Mateus Tremea1, Suellen Susin Gazzola2, Suele Bueno dos Santos3, Gadriéli Cristina gheno4, Alexendra Moller Alves5, Vanessa Seidel6, Rafael Aldrighi Tavares7, Heden Luiz Marques Moreira8
1 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
2 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
3 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
4 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
5 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
6 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
7 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
8 - Universidade Federal de Pelotas UFPEL
2 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
3 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
4 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
5 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
6 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
7 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
8 - Universidade Federal de Pelotas UFPEL
RESUMO -
Objetivou-se identificar o polimorfismo da região mitocondrial Citocromo b da espécie Hemisorubim phatyrhynchos. O programa Bowtie2 foi utilizado para realizar a montagem de referência, o gene mitocondrial utilizado como referência foi o da espécie Hemisorubim phatyrhynchos. Um total de 1065 reads foram alinhados a 1223 pares de base do gene de referência, revelando 49 sítios de diferenciação entre as espécies. A partir do sequenciamento de nova geração foi identificado o polimorfismo do gene mitocondrial Citocromo b da espécie Hemisorubim phatyrhynchos, sendo de grande relevância para base de trabalhos futuros de conservação e melhoramento genético.
Palavras-chave: Citocromo b; DNA mitocondrial; Jurupoca.
POLYMORPHISM OF CYTOCHROME B MITOCHONDRIAL GENE ON JURUPOCA SPECIES (Hemisorubim phatyrhynchos).
ABSTRACT - The objective of the study was to identify the polymorphism of the Cytochrome b mitochondrial region on the Hemisorubim phatyrhynchos. The Bowtie2 program was applied to do the reference assembly and the mitochondrial gene used as reference was the Hemisorubim phatyrhynchos. A number of 1065 reads was aligned to 1223 base pairs of the reference gene, revealing 49 differentiation sites between the species. From the sequencing of the new generation, the polymorphism of the Cytochrome b mitochondrial gene on Hemisorubim phatyrhynchos was identified, having great relevance as a basis for future works on conservation and genetic improvement.Keywords: Cytochrome b, mitochondrial DNA, Jurupoca.
Introdução
Hemisorubim platyrhynchos, popularmente conhecida como jurupoca, é uma espécie de médio porte, pertencente à família Pimelodidae e tem ocorrência nos grandes rios da América do Sul, nas bacias dos rios Amazonas, Maroni, Orinoco e Paraná (REIS et al., 2003). O genoma mitocondrial dos animais é, na grande maioria dos casos, constituído por uma molécula de DNA circular, pequena, com conteúdo gênico conservado (apenas 37 genes) e estrutura gênica simples (não possui DNA repetitivo) (Wilson et al., 1985). Essa extrema economia de conteúdo da maioria dos genomas mitocondriais animais é atribuída a uma intensa seleção a favor de um genoma pequeno (Moritz et al., 1987). Por outro lado, essa molécula apresenta alta taxa de evolução por mutação (Brown, 1985), cerca de 5 a 10 vezes mais rápida do que a taxa de mutação de um gene nuclear de cópia única (Moritz et al., 1987). A combinação dessas características faz com que o DNAmt seja amplamente utilizado em estudos de caracterização de populações, subespécies e espécies, além de estudos de caráter evolutivo e filogenético(Harrison, 1989). Com este trabalho objetivou-se identificar o polimorfismo do gene mitocondrial CYTB da espécie Hemisorubim platyrhynchos, relevando a variabilidade existente no indivíduo, e consequentemente, na espécie.Revisão Bibliográfica
A espécie jurupoca é predadora de microfauna bentônica e de peixes, com uma longevidade de 11,4 anos, podendo alcançar até 64 cm de comprimento (PENHA et al., 2003). Trata-se de uma espécie carnívora que alimenta-se de peixes e invertebrados, sua carne é de tonalidade amarelada de excelente sabor, sendo muito apreciada. O DNA mitocondrial (mtDNA) possui, a vantagem de ser muito conservado ( conteúdo e ordem dos genes) e de possuir maior taxa evolutiva ( taxa de substituição de bases) quando comparado ao DNA nuclear (nDNA). O fato de possuir herança somente materna na maioria das espécies faz com que esses marcadores sejam eficientes para acessar padrões de migração e relações e diferenças taxonômicas entre populações de uma mesma espécie. Por outro lado, isso pode se tornar uma desvantagem, pois esses marcadores só são capazes de traçar a linhagem materna (Arias et al,. 2003). Os marcadores mitocondriais são muito uteis para estudos populacionais, filogenéticos, filogeográficos e de aspectos biológicos e evolutivos em diversos organismos (Wilson et al,. 1985; Avise et al,. 1987; Moritz et al,. 1987; Arias et al,. 2003). O citocromo b que é a subunidade catalítica central da ubiquinol citocromo c reductase, uma enzima que está presente na cadeia respiratória da mitocôndria e na cadeia respiratória do ciclo foto-redox de muitas bactérias (MEYER, 1994), todos os organismos eucariotos necessitam desta classe de enzima redox, e consequentemente do citocromo b para a conversão de energia (ORREGO, 2012). É possível acessar informações e assegurar o conhecimento sobre populações naturais através do estudo genético molecular. Frankham et.al.2008.Materiais e Métodos
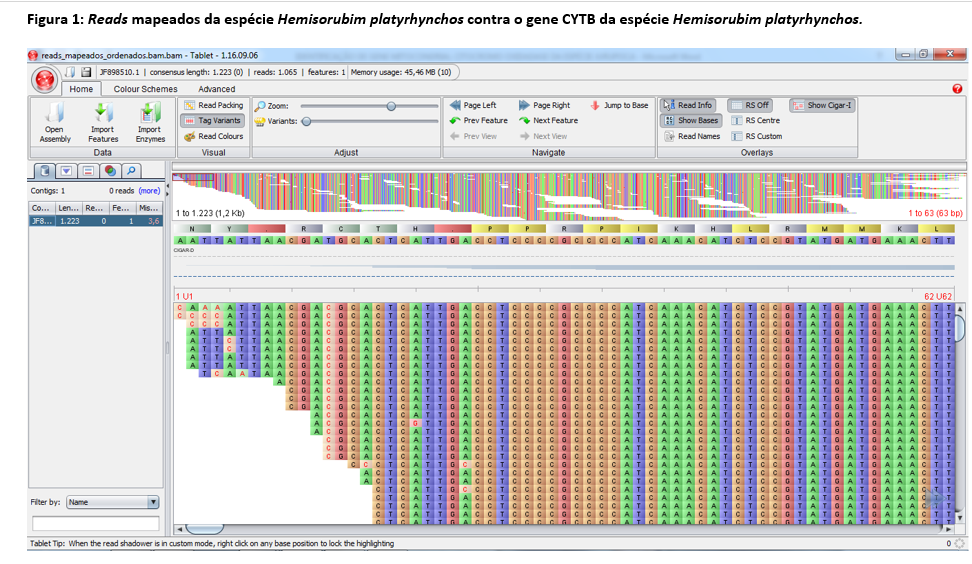
A biblioteca de DNA, da espécie Hemisorubim platyrhynchos foi obtida a partir de um sequenciador GAIIx (Illumina, USA) no modo paired-end, para a obtenção de sequências com 150 pares de bases (pb). A qualidade de cada read foi analisada com o programa FastQC (PATEL e JAIN, 2012). Após a análise de qualidade foi realizada a remoção dos adaptadores e a remoção de read de baixa qualidade com o programa Trimmomatic (BOLGER et al., 2014). As sequências das extremidades dos reads foram removidas quando as médias de qualidade fossem inferiores a Phread 15 em intervalos de quatro bases. Também foram removidos os reads com comprimentos menores que 32pb. Para a verificação da eficiência da filtragem foi utilizado novamente o programa FastQC. A montagem por referência foi realizado com o programa Bowtie2 (Langmead e Salzberg, 2012) sendo utilizado como referência o gene mitocondrial Citocromo b (GenBank: JF898510.1) com comprimento de 1223 pb da espécie Hemisorubim platyrhynchos. O arquivo no formato SAM, gerado na montagem, foi transformado em formato BAM com o programa Samtoll (LI et al., 2009) e posteriormente a reads montadas contra a referência foram visualizadas com o programa Tablet (MILNE et al., 2013).Resultados e Discussão
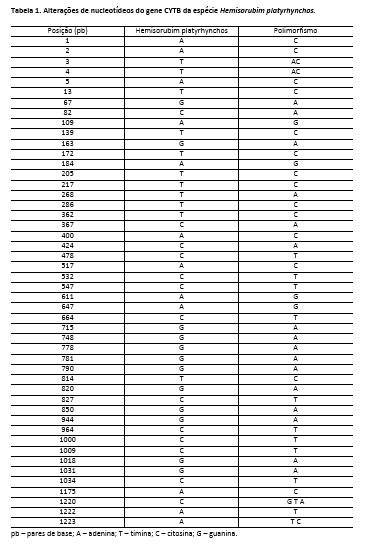
O sequenciamento de DNA resultou em aproximadamente 1.816.200 reads, sendo estes de 35 a 151pb, subdivididos em dois arquivos paired-end. Após foi realizada a filtragem através do programa Trimmomatic mantendo-se 1.762.764 reads e sendo removidos 53.436 reads. As bases que restaram apresentam-se de boa qualidade, com valores de Phred acima de 26. Segundo Del Fabbro et al. (2013), ocorre o aumento das taxas de alinhamento dos reads a um genoma de referência, mesmo com a diminuição do volume de sequências após a filtragem. Houve um total de 1065 reads alinhados aos 1223 pares de base do gene de referência CYTB da espécie Hemisorubim platyrhynchos (figura 1). Ocorreram 48 sítios de polimorfismo (tabela 1). Segundo ORREGO (2012) o uso do citocromo b se justifica pela presença de regiões conservadas e variáveis, as quais contêm sinais que podem ser utilizados em análises filogenéticas.Conclusões
A partir do sequenciamento de nova geração identificou-se o polimorfismo do gene mitocondrial Citocromo b da espécie Hemisorubim platyrhynchos. É de grande relevância caracterizar regiões mitocondriais para base de trabalhos futuros de conservação e melhoramento genético.Gráficos e Tabelas