SOFTWARE NIH IMAGEJ COMO UM POTENCIAL SISTEMA DE QUANTIFICAÇÃO PARA CARNES MARMORIZADAS
2 - Universidade Federal de Viçosa
RESUMO -
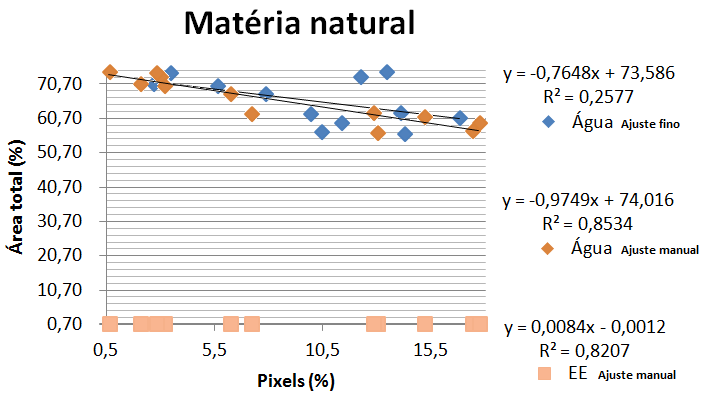
Desde que os equipamentos de imagem digital entraram para a ciência, pesquisadores buscam técnicas de processamento de imagem para criar biométria. Porém, as peculiaridades do processamento exigem adaptações específicas, as quais geram novos desafios de pesquisa. Este estudo visa verificar a efetividade do software NIH ImageJ para analisar informações visuais e quantificar dados em peças de carnes marmorizadas. Usamos comparações entre software, colorímetro, análises bromatológicas e estatística para verificar a confiabilidade dos dados. Obtivemos baixa correlação (R² 0,4122) no primeiro tratamento devido à interferência luminosa e boa no segundo (R² 0,8207), evidenciando que informações adicionais sobre área poderiam contrabalancear a nula informação sobre profundidade. O estudo conclui viabilidade (P<0,1) na criação de Macros para quantificação de dados em carnes marmorizadas.
NIH IMAGEJ SOFTWARE AS A POTENTIAL QUANTIFICATION SYSTEM FOR MARBLED MEATS
ABSTRACT - Since digital imaging equipment has entered science, researchers are looking for image processing techniques to create biometrics. However, the peculiarities of processing require specific adaptations, which generate new research challenges. This study aims to verify the effectiveness of NIH ImageJ software to analyze visual information and quantify data in pieces of marbled meats. We used comparisons between software, colorimeter, bromatological and statistical analyzes to verify the reliability of the data. We obtained a low correlation (R² 0,4122) in the first treatment due to light interference and good in the second (R² 0.8207), evidencing that additional information about area could counterbalance the null depth information. The study concludes the feasibility (P <0.1) in the creation of Macros for quantification of data in marbled meats.Introdução
Desde que os equipamentos de imagem digital entraram no mundo da ciência, biólogos colaboram com programadores para aplicar técnicas de processamento de imagem e analisar dados biométricos. A intenção é utilizar processos computacionais para acelerar tarefas repetitivas e obter resultados quantitativos, uma vez que os resultados estatísticos são muito mais atraentes do ponto de vista científico do que as observações qualitativas (J. Schindelin ET AL., 2015). Porém, as peculiaridades do processamento e tecnificação exigem adaptações específicas, as quais geram novos desafios de pesquisa e oportunidades, vista a crescente quantidade de publicações na área (LÉZORAY et al., 2012). O presente estudo visa verificar a efetividade e adaptabilidade do software NIH ImageJ para analisar informações visuais que, posteriormente poderão ser utilizadas em modelos matemáticos padronizados ou na criação de Macros para quantificação de dados em peças de carnes marmorizadas.Revisão Bibliográfica
O uso dos conceitos de Zootecnia de precisão requer alguns princípios importantes durante sua implantação, dentre os quais podemos destacar: obtenção contínua de informações do processo de resposta, podendo a saída de informações serem expressas em terminologias da engenharia ou em frequências e escalas adequadas ao feedback do processo controlador (Chizzotti et al., 2013). Segundo Mascarenhas e Velasco (1988), durante o processo de análise e identificação de imagens, é necessário a extração de medidas, características ou informação de uma dada imagem. Para isso, pode ser utilizada a técnica de segmentação, que consiste na subdivisão da imagem em partes ou objetos constituintes, permitindo a identificação de diferenças entre duas ou mais partes de um mesmo objeto, a discriminação de suas partes e a separação do objeto e o plano de fundo da imagem. Esta técnica possibilita a extração de características importantes da imagem, tais como bordas, texturas, cores ou métrica. Posteriormente, efetua-se a classificação das imagens a partir das características e representações obtidas pelas etapas anteriores do processamento. Com a coleta de imagens realizadas de forma adequada, podem-se analisar os índices que posteriormente serão utilizados em modelos matemáticos, padronizados para cada categoria. Estes mesmos índices, poderão ser correlacionados entre si através de equações matemáticas aleatórias, onde serão em outra etapa analisados estatisticamente, e de acordo com a relevância biométrica, podem ser escolhidos ou descartados (Lézoray et al., 2012).Materiais e Métodos
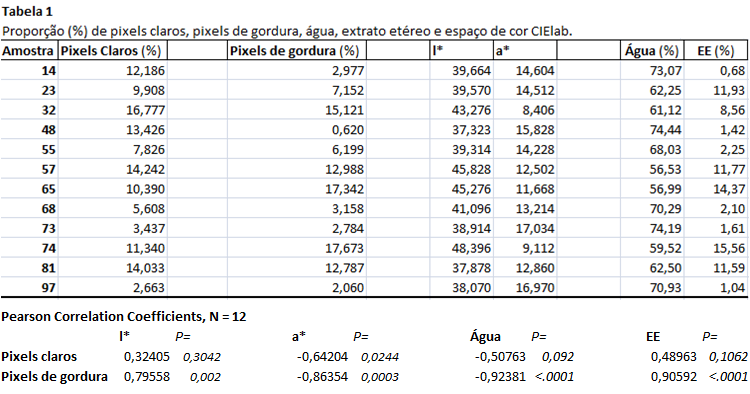
As 12 amostras de bifes marmorizados utilizadas no estudo foram doadas pelas empresas Intermezzo Gourmet, Beef Pasion e Swift e a determinação da composição química do bife foi realizada no Laboratório de Nutrição Animal da Universidade Federal de Viçosa. Os bifes foram pesados em fresco, liofilizados e parcialmente desengordurados através de lavagens sucessivas com éter de petróleo, obtendo-se a matéria seca pré-desengordurada (MSPG), e posterior moagem. Foram feitas análises de matéria seca (MS) e extrato etéreo (EE), seguindo a metodologia do INTC-CA descrita por Detmann et al. (2012). A determinação dos parâmetros L*, a* e b* usou o sistema CIE L*a*b* com o colorimetro Minolta CR-400 (Konica Minolta, Osaka, Japan), onde o L* é relativo a luminosidade (L* 0 = preto e 100 = branco) , a* é o índice que varia de verde (-) para vermelho (+) e b* é o índice que varia de azul (-) para amarelo (+) (Houben, Van Dijk, Eikelenboom, & Hoving-Bolink, 2000). O software ImageJ de licensa aberta desenvolvido pela National Institutes of Health (NIH), foi usado para outras análises de imagem. Neste, foram feitos dois tratamentos: ajuste fino e ajuste manual, objetivando comparação entre a acurácia do programa e a de um operador, que poderia ser transformada em um macro posteriormente. Para o ajuste fino foram usados valores que variam do 0 ao 255 no eixo de luminosidade do sistema HSB (hue, saturation e brightness), encontrado em Image>Adjust> Color Threshold. O ajuste manual foi feito contornando a gordura marmorizada encontrada nas amostras com posterior calculo das somas das áreas (analyze>measure). Para obtenção dos dados, todas as amostras tiveram suas áreas padronizadas em porcentagem (cm²/cm²) e unidade de medida calibrada em pixels por centímetros. Análises estatísticas, correlações, regressão e gráficos foram feitas usando SAS (SAS Inst. Inc., Cary, NC) e o software Microsoft Office Excel 2007.Resultados e Discussão
As medidas percentuais de pixels obtidas pela analise de imagens com o software ImageJ, os índices de cores obtidos por colorímetro e as estimativas das analises bromatológicas estão representados na tabela 1 e no gráfico 1. As correlações entre pixels e outros parâmetros foram baixas para ajuste fino (R² 0,2577 a 0,4122) e altas para ajuste manual (R² 0,6330 a 0,8207) no software. Tal variação pode ser explicada pela inadequada exposição da amostra à luminosidade no momento de coleta das imagens, resultando em erros de identificação dos pixels de gordura pelo software e assimilação de pixels brilhantes. Isto mostra que para o desenvolvimento de equações e macros responsáveis por predizer a porcentagem de gordura através de imagens, são necessárias condições adequadas de coleta para que não haja interferências luminosas. As análises estatísticas também revelaram relações entre os pixels analisados em software, os parâmetros capturados pelo colorímetro e as estimativas bromatológicas ao nível de 10% de significância. Tais fatos são devidos à sabida correlação negativa entre água e gordura: quanto mais pixels claros ou de gordura, menor a proporção de água na amostra (R²=0,8534 e P<0.0001). Curiosamente, a análise em duas dimensões também obteve boa correspondência com o extrato etéreo estimado em laboratório (R²=0,8207 e P<0,0001), evidenciando que a nula informação sobre profundidade poderia de alguma forma ser contrabalanceada com adicionais informações sobre área, satisfazendo as duvidas iniciais e criando expectativa sobre possíveis macros.Conclusões
Neste estudo, nós demonstramos a viabilidade do software NIH ImageJ na percepção e quantificação de dados em carnes marmorizadas. Foram feitos dois tratamentos para avaliação e diferenciação entre padrões automáticos e manuais de análises visuais usando o software, o que evidenciou necessidade (intensificada por interferências luminosas), mas também prosperidade na criação de novos macros que facilitem o estudo da ciência das carnes. Também ocorreram boas correlações entre as informações visuais analisadas manualmente, as capturadas por colorímetro e as estimadas em laboratório por análise bromatológica, o que realça promissor desenvolvimento desse nicho na área.Gráficos e Tabelas