AMPLIFICAÇÃO CRUZADA DE MARCADORES MOLECULARES MICROSSATÉLITES PARA Pseudoplatistoma reticulatum
2 - universidade estadual de maringá
3 - universidade estadual de maringá
4 - universidade estadual de maringá
5 - universidade estadual de maringá
6 - universidade estadual de maringá
7 - universidade estadual de maringá
8 - universidade estadual de maringá
RESUMO -
Os marcadores microssatélites (SSR’s) atualmente são referência no monitoramento genético de populações de peixes, contudo quando se trabalha com espécies não sequenciadas, sua utilização é dificultada pela inexistência de primers espécie-específico. O objetivo deste trabalho foi testar a amplificação cruzada de SSR’s originalmente desenvolvidos para P. corruscans, P. punctifer, B. rousseauxi em amostras de cachara (P. reticulatum). Dos 15 loci microssatélites testados, cinco não apresentaram amplificação satisfatória (fragmentos inespecíficos ou alelos monomórfico), enquanto que os restantes apresentaram amplificação nítida e polimórfica, evidenciando a eficácia dos primers utilizados. As sequências produziram total de 32 alelos, que variaram entre 105pb e 280pb. Conclui-se que a amplificação realizada com sucesso de 10 primers heterólogos para o cachara contribuirá de maneira significativa para a realização de novos estudos relacionados à genética populacional desta espécie.
CROSSED AMPLIFICATION OF MICROSSATELLITE MOLECULAR MARKERS FOR Pseudoplatistoma reticulatum
ABSTRACT - Microsatellite markers (SSRs) are currently a reference in genetic monitoring of fish populations, however when working with non-sequenced species their use is hampered by the lack of species-specific primers. The objective of this study was to test the cross-amplification of SSRs, originally developed for P. corruscans, P. punctifer, B. rousseauxi on samples of cachara (P. reticulatum). From the 15 microsatellite loci tested, five did not present satisfactory amplification (non-specific fragments or monomorphic alleles), while the remaining 10 showed clear and polymorphic amplification, evidencing the efficacy of the primers tested. The sequences produced a total of 32 alleles, ranging from 105bp to 280bp. It is concluded that the successful amplification of 10 heterologous primers for cachara will significantly contribute to the accomplishment of new studies related to population genetics of this species.Introdução
A técnica de PCR surgida nos anos 80, promoveu grande revolução em estudos genéticos, já que possibilitou a análise de um grande número de indivíduos de maneira rápida e prática. Desde então foram desenvolvidos uma série de marcadores moleculares capazes de revelar polimorfismos e que permitem a avaliação genética de indivíduos, comunidades e populações. Atualmente os marcadores microssatélites (SSRs) se tornaram referência no monitoramento genético de populações de peixes (Lopera-Barrero, 2007). Os SSRs são compostos por sequências de 1 a 8 nucleotídeos repetidos em tandem, muito abundantes no genoma eucarioto, e distribuídos ao acaso, permitem ampla cobertura. Essas características possibilitam a análise do comprimento de cada alelo herdado, facilitando a identificação de indivíduos homo e heterozigotos (Chistiakov et al., 2006). Embora sejam eficazes, quando o foco do estudo são espécies não sequenciadas, como grande parte dos peixes nativos brasileiros, esse processo é dificultado, já que primers espécie-específico são inexistentes e sua confecção representa um processo oneroso. A constatação da ocorrência de conservação de sítios em genomas de espécies relacionadas, esse problema foi resolvido, tornando possível em alguns casos, a amplificação cruzada de primers heterólogos. (Mia, 2005). O objetivo deste trabalho foi testar a amplificação cruzada de SSR’s originalmente desenvolvidos para P. corruscans, P. punctifer, B. rousseauxi em amostras de cachara (P. reticulatum).Revisão Bibliográfica
A descoberta da técnica de PCR na década de 80, desencadeou uma verdadeira revolução nos estudos genéticos, passando a ser possível a análise de um grande número de indivíduos de maneira rápida e prática. Esta técnica baseia-se na síntese in-vitro de milhões de cópias de um segmento específico de DNA, realizada através da ação da enzima DNA polimerase e permitindo a análise de variabilidade dentro e entre populações, com base nas diferenças de sequências de bases de segmentos no DNA (Ribolli, 2007). A partir daí, foram desenvolvidos uma série de marcadores moleculares, que permitiram a avaliação genética de indivíduos, comunidades ou populações. Os marcadores moleculares microssatélites são atualmente os mais utilizados em estudos genéticos de peixes, se tornando referência no monitoramento de estoques em cativeiro e populações naturais (Lopera-Barrero, 2007). Esses marcadores também conhecidos como sequência simples repetida (SSRs), são compostos por sequências de um a oito nucleotídeos repetidos em tandem. Esse tipo de marcador é o mais abundante no genoma eucarioto, e distribuídos ao acaso, permite sua ampla cobertura. Essas características possibilitam a verificação do comprimento de cada um dos alelos herdados, facilitando a identificação de indivíduos homo e heterozigotos (Chistiakov et al., 2006). Embora os SSRs apresentem excelentes resultados, quando o foco do trabalho são espécies ainda não sequenciadas, como a maioria dos peixes nativos brasileiros, esse processo é dificultado, já que muitas vezes os primers espécie-específico são inexistentes e sua confecção representa um processo caro e demorado. Com a constatação da ocorrência da conservação de sítios em genomas de espécies relacionadas, esse problema foi resolvido, tornando possível em alguns casos, a transferência de marcadores entre espécies ou gêneros, observando-se maior transferibilidade de loci heterólogos, quanto mais próximo forem as espécies taxonomicamente. (Mia, 2005).Materiais e Métodos
Cortaram-se amostras de nadadeira caudal de 48 animais a partir de um estoque de reprodutores de cachara da região de Cuiabá-MT. As amostras foram armazenadas em álcool 70% e analisadas no laboratório de Biologia Molecular do grupo de pesquisa Peixegen (UEM). Para a extração do DNA utilizou-se a metodologia descrita por Lopera-Barrero (2008). Os fragmentos de nadadeiras (0,5 cm2) foram colocados em microtubos, aos quais adicionaram-se tampão de lise, SDS e proteinase K. As amostras foram incubadas em banho-maria a 50ºC overnight. Posteriormente, o DNA foi purificado com NaCl, centrifugado por 10min a 12.000 rpm, e em seguida precipitado com etanol absoluto. O DNA precipitado foi lavado com etanol 70%, re-suspendido em TE e tratado com RNAse em banho-maria (38ºC por 1h). O DNA foi quantificado por espectofotometria e padronizado a 10nM/μl, com teste de integridade realizado em gel de agarose 1%. Inicialmente os testes de amplificação foram realizados com cinco amostras, e repetidos com o restante das amostras ao serem encontrados locus com amplificação nítida. A reação de amplificação foi realizada de acordo com especificações do fabricante da enzima TAQ (Uniscience, Miami, USA). Foram avaliados 15 primers heterólogos sendo suas informações são encontradas na tabela 1. As amostras amplificadas foram submetidas à eletroforese em gel de poliacrilamida desnaturante a 10% por sete horas. Para a visualização dos alelos microssatélites, foi utilizada coloração com nitrato de prata e fotografado com digital comum para análises posteriores. O tamanho dos alelos foi calculado pelo programa Kodak EDAS-290, utilizando-se DNA ladder (Invitrogen) de 10, 50 e 100 pb.Resultados e Discussão
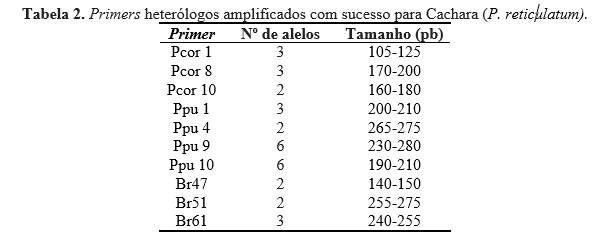
Dos 15 loci microssatélites testados, cinco não apresentaram amplificação satisfatória com fragmentos inespecíficos ou alelos monomórfico, foram eles Pcor5, Ppu2, Ppu15, Br38 e Br40. Este resultado pode ser explicado por Sun et al. (2012) que relataram que a amplificação cruzada de locus microssatélites está relacionada diretamente com a distância filogenética entre as espécies, sendo que a técnica oportuna em espécies do mesmo gênero, e menos eficiente entre as famílias ou gêneros diferentes. No entanto, os testes podem ser aplicados entre amostras de diferentes taxonomias e apresentar resultados aceitáveis, de modo que se utilize primers entre as espécies de peixes, o que foi observado para os três primers amplificados com sucesso. Formiga et al. (2010) por exemplo, testaram 36 primers microssatélites desenvolvidos para a Dourada (Brachyplatystoma rousseauxii) em amplificação cruzada para seis espécies do mesmo gênero e obtiveram 17 locus amplificados com sucesso em todas as espécies. Por outro lado, os dez primers restantes apresentaram amplificação nítida e polimórfica (Pcor1, Pcor8, Pcor10, Ppu1, Ppu4, Ppu9, Ppu10, Br47, Br51 e Br61). As informações dos primers amplificados com sucesso são apresentadas na Tabela 2. As sequências produziram um total de 32 alelos, que variaram entre 105pb e 280pb. Os alelos encontrados foram semelhantes aos encontrados por Naturae (2011) no Programa de Conservação da Ictiofauna da barragem hidroelétrica de jirau. Um resultado interessante foi que mesmo tendo sido amplificados primers heterólogos, os alelos ficaram extremamente nítidos, evidenciando a eficácia dos primers utilizados. Cabe ser ressaltado que as temperaturas de anelamento variaram para cada primer, fato que deve ser levado em consideração para repetibilidade de estudos futuros.Conclusões
Conclui-se que a amplificação realizada com sucesso de 10 primers heterólogos para o cachara (P. reticulatum), amplificação nítida e polimórfica, contribuirá de maneira significativa para a realização de novos estudos relacionados à genética populacional desta espécie.Gráficos e Tabelas