Análise de endogamia em ovinos de corte da raça Santa Inês
2 - Unesp/FCAV
3 - Unesp/FCAV
4 - IZ, Centro APTA Bovinos de Corte, Sertãozinho
5 - Unesp/FCAV
RESUMO -
O objetivo deste trabalho foi avaliar os níveis de endogamia em uma população de ovinos de corte da raça Santa Inês. A estrutura do pedigree foi analisada utilizando o programa Endog. O coeficiente de endogamia médio calculado para a população foi igual a 0,0091. O aumento na taxa de endogamia, para geração máxima, geração completa e geração equivalente foi igual a 0,62%, 1,62% e 1,18%, respectivamente. Devido ao aumento do coeficiente médio de endogamia por geração, recomenda-se controlar os acasalamentos a fim de estabilizar ou diminuir a endogamia média nas próximas gerações.
Inbreeding analysis in Santa Ines sheep
ABSTRACT - The aim of this study was to evaluate the levels of inbreeding in a Santa Inês sheep population. The structure of the pedigree was analyzed using the program Endog. The mean inbreeding coefficient calculated for the population was 0.0091. The rate of inbreeding per maximum generation, complete generation and equivalent generation was 0.62%, 1.62% and 1.18%, respectively. It was recommended to control the mating of the animals in order to stabilize or decrease the average inbreeding in the next generations, due to the increase in the average inbreeding coefficient per generation.Introdução
Endogamia é a probabilidade de dois alelos do mesmo locus serem idênticos por descendência (WRIGHT, 1922). O aumento da endogamia pode resultar no aumento da frequência de alelos indesejáveis na população (PEREIRA, 1999), resultando em depressão endogâmica perda de variabilidade genética e ainda pode afetar o desempenho fenotípico para certas características, principalmente reprodutivas (FALCONER; MACKEY, 1996). O controle dos acasalamentos entre animais aparentados é de grande importância para a obtenção do progresso genético das características que são objetivos de seleção no programa de melhoramento. Portanto, este trabalho teve como objetivo analisar os níveis de endogamia em uma população de ovinos de corte da raça Santa Inês a fim de monitorá-la na população.
Revisão Bibliográfica
O estudo da estrutura populacional, baseado em informações de pedigree, tem sido realizado em diferentes populações ovinas de diversas partes do mundo (MAIWASHE; BLACKBURN, 2004; TAHMOORESPUR; SHEIKHLOO, 2011; CERVANTES et al., 2011; SAFÁRI; JAMES, 2002; PAIVA et al., 2011; CHALH et al., 2012). Teixeira Neto et al. (2013) estudando ovinos da raça Santa Inês encontrou coeficiente de endogamia de 0,0692, e concluiu que este coeficiente requer monitoramento por sua proximidade do limite recomendável e que o fluxo de genes entre os rebanhos é o principal fator para o aumento do tamanho efetivo e a manutenção da variabilidade genética da raça Santa Inês.Materiais e Métodos
Os dados utilizados foram provenientes de informações de pedigree de 2.410 animais da raça Santa Inês, pertencentes a seis fazendas do estado de São Paulo, que possuíam ligação genética entre si. A análise do pedigree foi realizada a partir do software Endog (GUTIÉRREZ; GOYACHE, 2005). O coeficiente de endogamia, definido pela probabilidade de um indivíduo possuir dois alelos idênticos por descendência (WRIGHT, 1922), foi calculado por meio do algoritmo desenvolvido por Meuwissen e Luo (1992). O aumento da endogamia pelas gerações foi calculado a partir da equação: Δ????=(????????−????(t−????)/????−????(t−????)), sendo que ???????? e ????????−1 são a endogamia média na ????é???????????????? e ????é????????????????−1 geração (WRIGHT, 1922).Resultados e Discussão
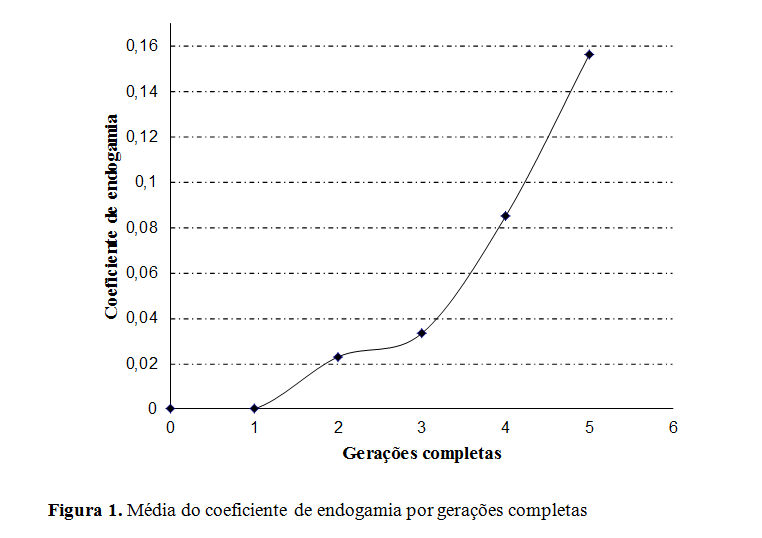
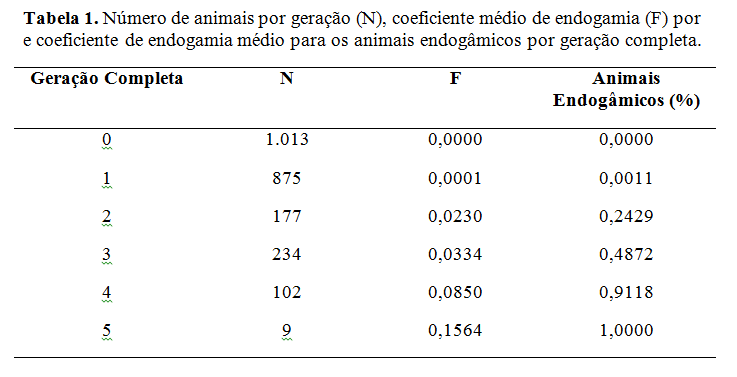
O coeficiente de endogamia médio calculado para a população foi igual a 0,0091 e sua variação ao longo das gerações completas está representada na Figura 1. O animal que apresentou maior coeficiente de endogamia foi uma fêmea, com 0,3125. O aumento na taxa de endogamia, para geração máxima, geração completa e geração equivalente foi igual a 0,62%, 1,62% e 1,18%, respectivamente. Os valores apresentados no aumento da taxa de endogamia para geração máxima foram menores se comparada com a geração completa e equivalente, pois há falta de informação no pedigree dos animais nas primeiras gerações, portanto esta estimativa pode estar subestimada. A ausência de endogamia verificada nas primeiras gerações pode ser pelo número reduzido de ancestrais conhecidos até então, e não pela ausência de animais endogâmicos na população, o que impossibilitou a estimação desse parâmetro. Foi observado aumento no coeficiente de endogamia ao longo das gerações (Tabela 1), assim como a porcentagem de animais endogâmicos.Conclusões
Devido ao aumento do coeficiente médio de endogamia por geração, recomenda-se controlar os acasalamentos a fim de estabilizar ou diminuir a endogamia média nas próximas gerações. Agradecimentos A FAPESP (financiamento do projeto: 2012/15982-0 e processos: 2016/10583-1 e 2013/20091-0), a CAPES e ao CNPq.Gráficos e Tabelas