Análise multivariada aplicada a características biométricas de ovinos em confinamento
2 - FMVZ/UNESP
3 - FMVZ/UNESP
4 - FMVZ/UNESP
5 - FMVZ/UNESP
6 - FMVZ/UNESP
7 - Universidade Federal de São João del-Rei (UFSJ)
8 - FMVZ/UNESP
RESUMO -
O objetivo do trabalho foi utilizar análise multivariada para estudar a relação entre as características de desempenho e biométricas de cordeiros. Foram utilizados 140 animais inteiros com média de idade de 45 dias que permaneceram em confinamento por 45 dias. Dados fenotípicos biométricos foram coletados por meio de medidas objetivas. Análise de variância foi realizada para verificar a influência dos efeitos de grupo genético e suas interações com as características analisadas, além de modelo de regressão simples e análise multivariada. Componentes principais (CP) obtidas por meio da matriz de correlação apresentaram 79,6% da variância total foi explicado em três CP. Conclui-se que peso inicial determina a maior parte da variabilidade e comprimento da garupa não contribui com a variabilidade das características biométricas analisadas. A seleção de animais com maiores alturas deve ser realizada com atenção devido ser antagônica à características diretamente relacionada com precocidade.
Multivariate analysis applied to biometric traits of feedlot sheep
ABSTRACT - The aimed of study was to use multivariate analysis to evaluate the relationship between performance and biometrics traits of lambs. We used 140 non-castrated lambs with average age of 45 days, that remaining in confinement for 45 days. Biometric phenotypic data were collected using objective measures. Variance analysis was performed to verify the influence of genetic group effects and their interactions with the analyzed traits, besides simple regression model and multivariate analysis. Principal components (PC) obtained through the correlation matrix showed 79.6% of the total variance was explained in three PC. It is concluded that initial weight determines most of the variability and, lenght croup does not contribute to the variability of the biometric traits analyzed. The selection of animals with higher heights should be performed with attention due to being antagonistic to the traits directly related to precocity.Introdução
Nas últimas décadas a ovinocultura no Brasil vem demonstrando alto crescimento. Em contrapartida a este crescimento, o consumo de carne ovina no Brasil ainda é baixo, frente ao consumo de carne de outras espécies. Esse distanciamento pode ser explicado pela falta de programas de melhoramento genético aliados a nutrição, sanidade e necessidade de novos recursos biotecnológicos aplicados a espécie. Diante disto, pesquisadores vêm utilizando uma ferramenta para analisar várias características conjuntamente em um mesmo indivíduo, na qual permite estudar populações diferentes quando todas as variáveis biométricas são consideradas um conjunto (PIRES, 2009). Esta análise é conhecida como análise multivariada. O objetivo da análise multivariada utilizando componentes principais (ACP) é explicar a estrutura de (co)variância de um vetor aleatório, composto de p-variáveis aleatórias, construídas de combinações lineares das variáveis originais. Essa combinação chamada de componentes principais não são correlacionados entre si (MINGOTI, 2007). Com isso, o objetivo do presente trabalho foi estudar a relação entre características biométricas e ganho de peso de cordeiros terminados em confinamento por meio da análise multivariada no intuito de criar um conjunto menor de variáveis obtidas como função das variáveis originais.Revisão Bibliográfica
A utilização de informações biométricas na ovinocultura tem como finalidade além de caracterizar fenotipicamente as raças, pode contribuir com elaboração de índices de seleção assim como predizer valor genético e fenotípico dos animais, reposta e sentido da correlação (fenotípica e genética) e avaliar o progresso alcançado com a seleção. Assim, é possível melhorar economicamente a produção de proteína de origem animal. Para tal, pode-se utilizar análises multivariadas que, segundo Biagiotti et al. (2014), é uma ferramenta que analisa diversas características de um único indivíduo ou população. Esta apresenta relações de similaridade e dissimilaridade, onde são expressas distâncias genéticas ou métricas entre características. Segundo Pires (2009), a análise multivariada engloba técnicas numéricas usadas para estudar e descrever a covariação entre variáveis. Esta análise possui diferentes métodos de realização, dependendo do tipo de hipótese gerada a partir dos dados coletados e pode-se com a mesma reduzir o campo de variáveis analisadas retendo assim apenas aquelas com maiores porcentagens de variância total (VICINI & SOUZA, 2005).Materiais e Métodos
O estudo foi conduzido durante o Campeonato Cordeiro Paulista 2016 realizado pela Associação Paulista de Criadores de Ovinos (ASPACO) no período de 23/09 a 07/11/2016 no município de Araçatuba, SP. Foram coletadas as características biométricas de 140 cordeiros não castrados de distintos grupos raciais, desmamados com 45 dias de idade e submetidos a confinamento até atingir idade média de 90 dias. Dentre os 140 cordeiros apresentaram-se animais de diferentes grupos genéticos: Ile de France, Cruza Ile de France, Cruza Dorper, Suffolk, Cruza Suffolk, Dorper x Santa Inês, Santa Inês, Cruza Santa Inês. Todos os cordeiros foram submetidos a mensurações do Comprimento do animal (CC), Profundidade do animal (PA), Circunferência torácica (CT), Altura do anterior (AA), Altura do posterior (AP), Peso inicial (PI), Comprimento de garupa (CG). A pesagem corporal foi realizada individualmente em balança (kg) e a morfometria corporal dos cordeiros foi obtida com o auxílio de fita métrica (cm), com o animal mantido em posição de aprumos, conforme metodologia sugerida por Yañez et al. (2004). Análise de variância foi realizada para verificar a influência dos efeitos de grupos genéticos e suas interações com as características analisadas. Para tal análise foi utilizado o procedimento PROC GLM do SAS (SAS 9.1, SAS Institute, Cary, NC, USA). O modelo de regressão simples, utilizando quadrados mínimos foi aplicado com a finalidade de analisar os efeitos referentes a genética dos animais, sendo que somente os efeitos com significância menor do que 5% (p<0,05) foram considerados nas análises. A análise multivariada foi realizada utilizando o procedimento PROC PRINCOMP do SAS (SAS 9.1, SAS Institute, Cary, NC, USA). Segundo REYS (1997), esta ferramenta estatística possibilita a criação de um conjunto menor de variáveis obtidas como função das variáveis originais. Para tal se utiliza dos autovalores a partir dos critérios estabelecidos por KAISER (1960), que baseia-se em incluir somente os componentes cujos valores próprios sejam superiores a 1 associados com variância acumulada total acima de 70%. Este critério tende a incluir poucos componentes quando o número de variáveis originais for inferior a 20 e, utiliza-se os componentes principais que conseguem sintetizar uma variância acumulada total em torno de 70%.Resultados e Discussão
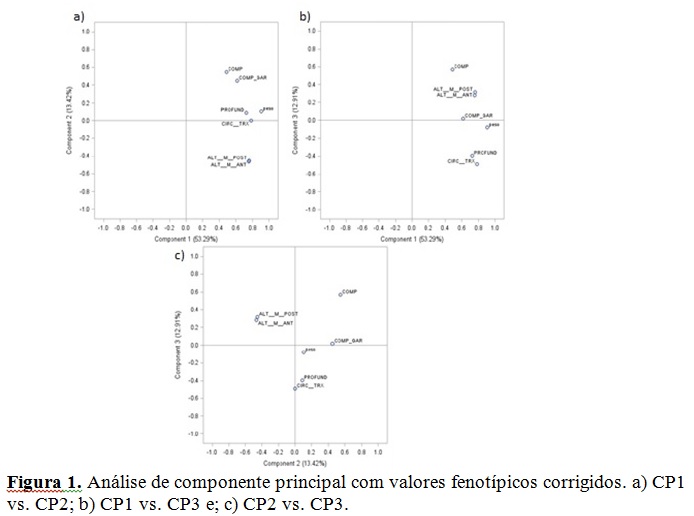
De acordo com os resultados obtidos por meio da análise estatística descritiva (Tabela 1), foi possível identificar variabilidade fenotípica dentro da mesma população devido os intervalos mínimo e máximo. Moreno et al. (2010), observaram médias superiores para características biométricas comparadas a do presente estudo. A possível variação e menores médias para as características estudadas, se deve ao fato da população ser heterogênea e serem provenientes de distintos grupos genéticos. Na análise de componentes principais (CP) obtida por meio da matriz de correlação foi possível obter 79,6% da variância total dos dados fenotípicos explicado em três CP. Esta porcentagem da variância total acumulada foi formada pelas proporções de 53,29% (CP1), 13,42% (CP2) e 12,91% (CP3). O total de variância se reflete principalmente quando se analisa os dois primeiros CP no qual, pela distribuição bidimensional do conjunto de características tem-se 66,71% da variação total. Em relação ao CP1 (Figura 1 a) todas as características apresentaram valores positivos, sendo o peso inicial a variável que mais contribuiu para a variação. Diferenças existentes são observadas quando se leva em consideração CP2 em que AA e AP não apresentam associação com CC e CG ou seja, maiores valores fenotípicos para AA e AP estariam associados com menores valores fenotípicos de CC e CG (Figura 1 a). Em relação aos resultado obtido na distribuição bidimensional entre CP1 e CP3 (66,20% da variância total), CC, AA e AP não apresentaram associação com as variáveis CT e PA (Figura 1 b). Foi observado que o PC1 foi uma dimensão que não discriminou o valor fenotípico para comprimento de garupa pois a projeção de COMP_GAR em PC1 ficou próxima do eixo (próximo de 0). Já ao comparar a distribuição entre CP2 e CP3 (26,33% da variância total) verificou-se alta associação entre AA e AP além de PA com CT (Figura 1 c). Esta associação demonstrou expressiva correlação positiva entre as variáveis. Entretanto a correlação é inversamente proporcional quando se observa as variáveis AA e AP comparadas a PA e CT, por estas estarem no quadrante negativo e as duas primeiras (AA e AP) no quadrante positivo.Conclusões
Conclui-se que o peso inicial determina a maior parte da variabilidade e que o comprimento da garupa não contribui com a variabilidade das características biométricas analisadas. Assim também se torna necessária atenção entre a seleção de animais com maiores alturas devido serem antagônicos a profundidade do animal diretamente relacionada com precocidade.Gráficos e Tabelas