Caracterização genética da sexta e sétima gerações de tilápia do Nilo do programa de melhoramento genético da Universidade Estadual de Maringá
2 - Universidade Estadual de Maringá
3 - Universidade Estadual de Maringá
4 - Universidade Estadual de Maringá
5 - Universidade Estadual de Maringá
6 - Universidade Estadual de Maringá
7 - Universidade Estadual de Maringá
8 - Universidade Estadual de Maringá
RESUMO -
A Universidade Estadual de Maringá (UEM) comporta um programa de melhoramento genético de tilápias em condições brasileiras de cultivo que demonstram o aumento da qualidade genética dos animais ao longo das gerações. O trabalho teve como objetivo caracterizar geneticamente, por meio de marcadores microssatélites, a sexta e sétima gerações do programa de melhoramento genético da UEM. Para tanto, foram avaliados 70 animais por geração a partir da amplificação de nove loci microssatélites. Os indicadores de diversidade genética se mostraram menores da sexta para a sétima geração, entretanto a análise de variância molecular e diferenciação genética indicaram a manutenção da variabilidade genética entre as gerações indicando que manutenção no programa de melhoramento genético da UEM tem sido eficiente.
Genetic characterization of the sixth and seventh Nile tilapia generations of genetic improvement program of the Universidade Estadual de Maringá
ABSTRACT - The State University of Maringá (UEM) involves a program of genetic improvement of tilapias in Brazilian conditions that demonstrate the increased genetic quality of animals along the generations. The work aimed to genetically characteriseby means of microsatellite markers, the sixth and seventh generations of genetic improvement program of UEM. To this end, 70 animals per generation were evaluated from the amplification of nine microsatellite loci. Genetic diversity indicators were lower from the sixth to the seventh generation, however molecular variance analysis and gene differentiation indicated mantenance of genetic variability among the generations indicating that maintenance in the genetic improvement program of UEM has been efficient.Introdução
A aquicultura é de importante representatividade quanto ao fornecimento de alimentos e segurança alimentar no mundo. Dentre a produção aquícola mundial os peixes representam a 48,8%, sendo a tilápia (Oreochromis spp) a espécie de segunda maior importância neste contexto (FAO, 2008). Para atender a demanda crescente, o melhoramento genético dos animais é fundamental, visto que indivíduos geneticamente superiores apresentam desempenho elevado em condições ambientais específicas, superioridade quanto à produtividade e a sobrevivência em relação a indivíduos outrora produzidos naquelas condições (De Resende et al., 2014) Em 2005, a Universidade Estadual de Maringá (UEM) recebeu tilápias da linhagem GIFT (Genetically Improved Farming Tilapia) com representantes de 30 famílias. Com isso, o Brasil tornou-se o primeiro país latino americano a receber linhagens provenientes de um programa de melhoramento genético. Desde então, nove gerações foram produzidas, e os resultados mostram aumento de 6% ao ano em ganho de peso diário. Tendo em vista a importância da variabilidade genética na condução de um programa de melhoramento genético o objetivo deste trabalho foi caracterizar geneticamente, por meio de marcadores microssatélites, a sexta e sétima gerações da tilápia do Nilo variedade TILAMAX, do programa de melhoramento genético da Universidade Estadual de Maringá.Revisão Bibliográfica
O Brasil pode figurar em breve entre os líderes mundiais de produção piscícola, já que possui cerca de 12% de todo o reservatório de água doce do planeta, além de mais de 8.000km de faixa marítima. (MPA, 2012). Dentre as espécies mais produzida a tilápia ocupa a segunda colocação, isto deve-se as suas boas características de produção como rápido crescimento, rusticidade, aceitabilidade do mercado consumidor, qualidade de carne e facilidade no filetamento (Moreira et al., 2007). Na Universidade Estadual de Maringá, há um programa de melhoramento genético com nove gerações formadas para a condição brasileira de cultivo, contudo, a prática do melhoramento genético deve ser realizada com cautela, levando-se em conta cuidados quanto a formação de reprodutores e do manejo reprodutivo famílias, uma vez que a utilização de um número reduzido de reprodutores, acasalamento de indivíduos aparentados e protocolos reprodutivos inadequados podem ocasionar perda de variabilidade genética nas futuras gerações, elevando os índices endogâmicos e diminuindo a produtividade. (Lopera-Barrero et al., 2014). Considerando a importância da variabilidade genética em um programa de melhoramento genético, uma técnica para avaliação desta é a utilização de marcadores moleculares, os quais permitem a estimativa dos coeficientes de endogamia e parentesco e desta forma a avaliação da relação genética entre gerações (Fessehaye et al., 2006). Dentre os marcadores moleculares, os marcadores microssatelites possuem algumas vantagens tais como a possibilidade da observação da codominância, abundância e natureza multialélica, além disso são facilmente detectados por PCR e possuem necessidade de pequenas quantidades de DNA para análise (Lima., 1998).Materiais e Métodos
Foram coletadas amostras da nadadeira caudal de 70 representantes de tilápias variedade TILAMAX, da sexta e sétima geração do programa de melhoramento da UEM em microtubos contendo álcool 70%. Para extração de DNA foi utilizada a metodologia descrita por (Lopera-Barrero et al., 2008). O DNA foi quantificado em espectrofotômetro PICODROP® e padronizado para concentração final de 10 ng/µL. A integridade do DNA foi verificada em gel de agarose 1%. Foram utilizados nove loci microssatélites descritos para a espécie (UNH 104, 140, 159, 160, 162, 163, 169, 190 e 231). A reação de amplificação foi realizada em termociclador Veriti® para um volume final de reação de 16mL, utilizando-se 1X do tampão Tris-KCl, 2,5 mM de MgCl2, 0,8 µM de cada primer (Forward e Reverse), 0,4 mM de cada dNTP, 1 U de Platinum Taq DNA Polimerase e 30 ng deDNA alvo. Os conjuntos de 10 µL dos produtos amplificados e 4 µL de tampão de corrida azul de bromofenol foram submetidos à eletroforese vertical em gel de poliacrilamida 10% por sete horas. Para a visualização dos alelos microssatélites, os géis foram corados com nitrato de prata segundo metodologia descrita por (Bassam et al.,1991), com modificações. O gel foi submetido a uma solução de fixação por 20 min, depois outra solução contendo 6 mM de nitrato de prata por 30 min e posteriormente, com uma prévia lavagem com água destilada, foi submerso numa solução reveladora. Os géis foram fotografados e o tamanho dos alelos obtidos foi estimado por comparação com marcadores de peso molecular de 100 pb (Invitrogen®, EUA). As análises de frequências alélicas, número e número efetivo de alelos, heterozigosidade observada e esperada, índice de fixação e equilíbrio de Hardy-Weinberg foram obtidos pelo programa estatístico PopGene 1.31, a identificação de alelos nulos pelo programa Micro-Checker e os valores de diferenciação genética e a análise de variância molecular pelo programa Arlequim 3.0.Resultados e Discussão
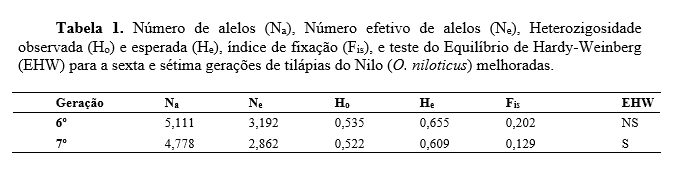
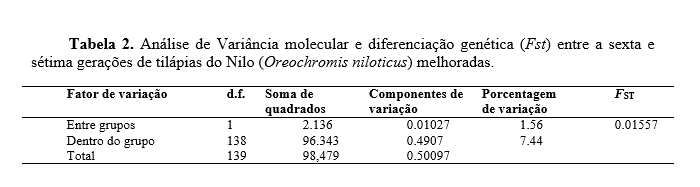
Os nove loci amplificados produziram 43 alelos, variando de quatro (UNH 140, 159, 190 e 231) a seis alelos por locus (UNH 160 e169). O tamanho dos alelos esteve entre 130pb (pares de base) (UNH 160) e 250pb (UNH 159). Os índices de diversidade genética intrapopulacionais são apresentados na tabela 1. Foram observadas diferenças entre o número médio e o número efetivo de alelos para as duas gerações, este fato se deve a existência de alelos nulos e a presença de alelos raros (p<0.05) ou com baixa frequência (0.05>p<0.25) nos loci estudados. Os valores de heterozigosidade observada (Ho) e heterozigosidade esperada (He), que representam o grau de diversidade genética dentro das gerações, diminuíram da sexta para a sétima geração. O coeficiente de endogamia (Fis) em ambas as gerações foi positivo, indicando a existência de um déficit de heterozigotos, sendo maior para a 6º geração. Esse déficit pode estar associado ao tipo de cruzamentos utilizados no programa de melhoramento. Segundo (Reis et al.,2011) valores positivos de Fis em programas de melhoramento são esperados, já que a intensidade de seleção pode ocasionar deriva genética, ao se reduzir o tamanho efetivo da população originada, dada pela recombinação das progênies selecionadas. Foi observado desvio no equilíbrio de Hardy-Weinberg na 7º geração, isto pode ser causado por diversos motivos, como: endogamia, mistura de populações com diferentes frequências de alelos, endocruzamento, excesso de homozigose e presença de alelos nulos (Kordicheva et al., 2010). No presente estudo, o desequilíbrio observado nos resultados pode ser explicado pelo endocruzamento na formação das gerações. A análise de variância molecular (Tabela 2) demonstrou baixa diferenciação genética entre as gerações, uma vez que a maior fonte de variação esteve dentro das gerações e não entre as gerações. Resultado corroborado pelo índice de fixação (Fst), que de maneira semelhante demonstrou baixa diferenciação genética.Conclusões
Conclui-se que mesmo com redução dos indicadores de diversidade genética (Na, Ne, Ho e He) as características de diversidade genética intra-populacionais, coeficiente de endogamia, equilíbrio de Hardy-Weinberg e índice de fixação indicaram a manutenção da variabilidade genética entre as gerações e que o critério para manutenção da variabilidade genética neste programa de melhoramento genético tem sido eficiente.Gráficos e Tabelas