Desenvolvimento de sistemas multiplex com marcadores microssatélites para um peixe de piracema ameaçado: Piracanjuba.
2 - Universidade Federal de São João del-Rei
3 - Universidade Federal de São João del-Rei
RESUMO -
Apresentamos a validação de multiplexes de microssatélites para Brycon orbignyanus, úteis para estudos e práticas de manejo. Esta espécie ameaçada é apreciada pelo sabor da carne, para pesca esportiva, tem bom valor comercial e bom potencial para pisciculturas. Multiplexes conciliam ensaios simultâneos de PCR. Os sistemas multiplex para B. orbignyanus foram desenhados a partir da avaliação de combinações de marcadores que geram faixas de fragmentos amplificados mutuamente exclusivas e sem formação de dímeros. Os sistemas foram desenvolvidos e padronizados em relação às condições da PCR. Foram obtidos quatro sistemas contendo combinação de três marcadores e dois sistemas com dois marcadores, com total de seis sistemas multiplex, englobando 16 microssatélites. Metade dos marcadores demonstrou equilíbrio de Hardy-Weinberg, após uma correção de Holm-Bonferroni. Esses sistemas aumentam em 2,5 vezes a eficiência da análise destes loci e serão úteis para sua conservação e piscicultura.
Development of multiplex systems with microsatellite markers for a threatened piracema fish: Piracanjuba.
ABSTRACT - We present the validation of microsatellite multiplexes for Brycon orbignyanus, useful for its conservation and management practices. This threatened species is appreciated for its flavor and for sports fisheries, with a good trade value and high potential for hatcheries. Multiplexes conciliate simultaneous PCR essays. The B. orbignyanus systems were designed based on mutually exclusive amplification ranges and heterodimer avoidance. The systems were developed and optimized regarding PCR conditions. Four systems with three markers and two with two markers were obtained, encompassing 16 microsatellites. Half the markers exhibited Hardy-Weinberg equilibrium, after a Holm-Bonferroni correction. These systems increase the efficiency of analyses of these loci in 2.5 times and will be useful in its conservation and hatchery initiatives.Introdução
Na agropecuária brasileira a aquicultura tem sido o setor com o maior crescimento segundo Food and Agriculture Organization (FAO, 2016). A espécie Brycon orbignyanus (Valenciennes, 1850), ou piracanjuba, é um peixe de piracema da família Bryconidae, que prefere águas claras e são normalmente encontrados em locais onde as árvores deitam sobre o rio, para se alimentar. A espécie já foi considerada como uma das principais para pesca esportiva e comercial na bacia do alto rio Paraná e hoje está praticamente extinto na natureza, sendo mantido, principalmente, por força de medidas de produção em pisciculturas voltadas para atividade de estocagem ambiental (peixamentos). A espécie oferece uma boa oportunidade para o desenvolvimento de novas frentes na aquicultura com peixes nativos e apresenta características favoráveis para produção em cativeiro. Diante destes desafios, o desenvolvimento de marcadores e tecnologias moleculares é de grande importância para o fomento das atividades de produção e conservação nesta espécie. Assim, propomos aqui, o desenvolvimento de reações multiplexadas de PCR com os primeiros marcadores moleculares tipo microssatélites isolados em B. orbignyanus, capazes de acelerar a caracterização de diversidade molecular de estoques e economizar insumos na produção de resultados.Revisão Bibliográfica
A piracanjuba é considerada em risco de extinção de acordo com o Livro Vermelho da Fauna Ameaçada de Extinção (Rosa e Lima, 2008). Na multiplex duas ou mais sequências alvo podem ser amplificadas com inclusão de dois ou mais pares de iniciadores em uma mesma reação. Essa PCR possui vantagens como a de economizar tempo e também ser menos laboriosa (Pereira, 2015). A FAO ressalva que várias são os motivos para a conservação dos recursos genéticos para alimentação e agricultura e que isto é imprescindível para conservar a adaptabilidade de cultivares e populações selvagens para preservar características genéticas que assegurem uma produção sustentável de alimentos (Hilsdorf, 2013). A PCR quantitativo fluorescente é uma técnica onde são utilizados iniciadores marcados com diferentes fluorocromos para amplificação da região alvo, sendo analisada através da eletroforese capilar, onde é possível analisar diversas sequências alvos próximas com mais pares de iniciadores (Delatorre, 2009). Essa técnica é caracterizada por sua precisão e sensibilidade como também ter uma resposta mais rápida comparada com a PCR tradicional. Foram realizadas avaliações com 73 amostras de Colossoma macropomum (tambaqui) utilizando reações multiplexadas resultando em boa capacidade de quantificar a variabilidade genética da população (Hamoy & Santos, 2012). Hamoy et al. (2008) descreveu o desenvolvimento de um painel multiplex de oito marcadores microssatélites para verificação de variabilidade genética em uma população natural de Arapaima (pirarucu) utilizando 32 amostras onde foi constatada uma média de 0,69, o número de alelos variou em 8 por locus, concluindo que esse painel pode ser utilizado como um método rápido e barato para avaliação da variabilidade genética. Cryer et al. (2005) descreveu uma abordagem simples e barata para selecionar marcadores microssatélites úteis utilizando iniciadores marcados por fluorescência em sistema capilar em organismos eucariotos. Para que o sistema multiplex tenha maior eficiência é necessário a utilização de muitos locis polimórficos e que não se sobrepõem a intervalos alélicos. Segundo Hilsdorf (2013) a manutenção da variabilidade genética é importante para a conservação dos estoques de peixes explorados pela pesca, essa manutenção é realizada através de estudos sobre as diferenças genéticas dentro e entre populações. Os peixes nativos advindos de piscicultura possuem um aumento na contribuição na comercialização de pescado.Materiais e Métodos
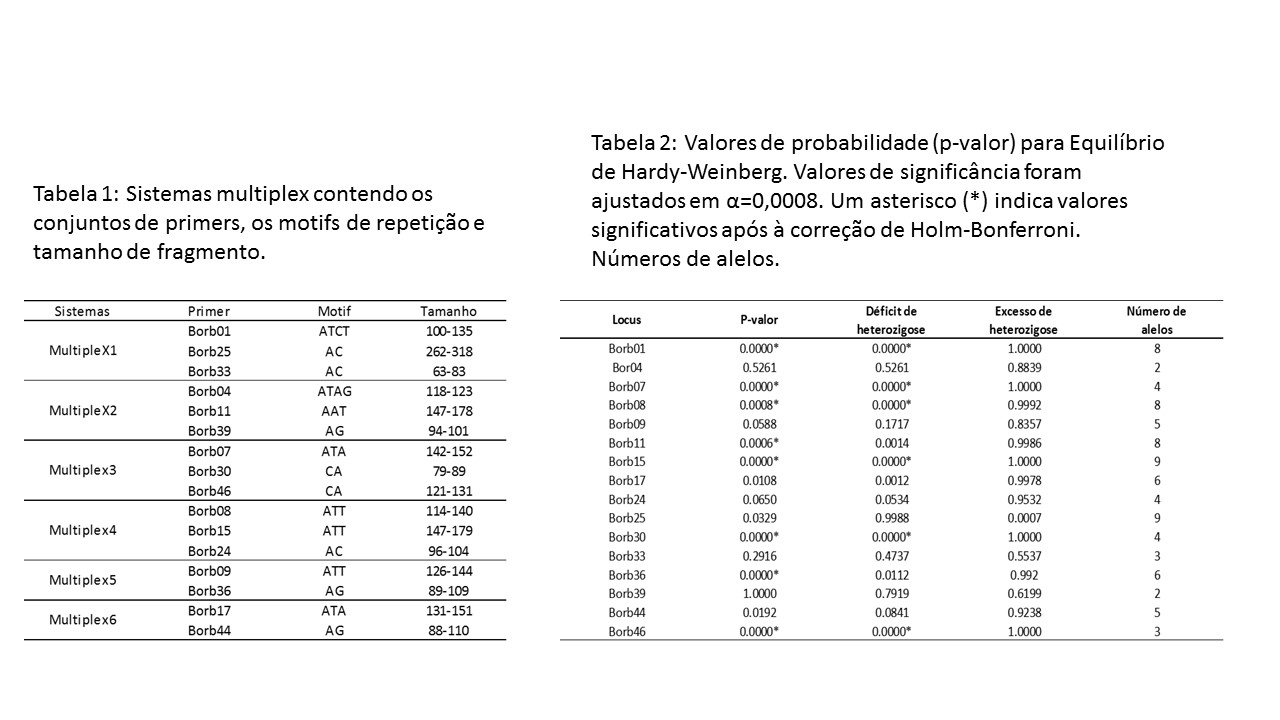
Foram utilizados 29 marcadores para Brycon orbignyanus validados no LARGE-UFSJ, descritos originalmente em Arias et al., (2016). Foram realizadas avaliações nos marcadores quanto a combinações de pares de primers que geram faixas de fragmentos amplificados que sejam mutuamente exclusivas e sem formação de dímeros. A avaliação de dímeros foi realizada com Primer3 (Untergasser et al., 2012). Em colaboração com o bolsista do LARGE, José Mauro Ribeiro, foi desenvolvido um algoritmo denominado Helpex para avaliação automática das combinações de pares de primers para os possíveis sistemas para multiplex, baseado na teoria dos grafos. Com os possíveis sistemas selecionados, foram feitos testes de gradiente de temperatura de anelamento de todos os marcadores para otimização de apenas uma temperatura em que todos pudessem ter padrões de bandas com boa qualidade. Em seguida foram realizadas as otimizações das reações em cadeia da polimerase (PCR). Os produtos de PCR foram aplicados em géis de poliacrilamida (10%) e submetidas à eletroforese, para separação de fragmentos de DNA amplificados. Os géis foram fotografados para análise posterior dos padrões de bandas do multiplex. Os resultados foram agrupados em classes de diferentes alelos e foram analisados com GenePop (versão 4.1.4, Rousset, 2008), que testa a conformidade quanto ao Equilíbrio de Hardy-Weinberg (EHW), para cada locus. Valores de significância foram ajustados com um procedimento de Holm-Bonferroni para testes múltiplos. Os 13 marcadores moleculares não aproveitados no desenvolvimento dos sistemas multiplex, foram utilizados para desenho de sistemas multiplex para diferenciação baseados em fluorocromos e eletroforese capilar. Esses marcadores foram avaliados quanto o seu tamanho de fragmento e a formação de dímeros entre eles, para análise de formação de dímeros foi utilizado o Primer3 (Untergasser et al., 2012).Resultados e Discussão
Foram descritos nesse trabalho seis sistemas de multiplex utilizando 16 marcadores para Brycon orbignyanus os quais são apresentados na Tabela 1, tendo uma boa porcentagem de aproveitamento dos 29 marcadores utilizados, sendo 55%. Os quais foram submetidos a PCR multiplex populacional com 36 indivíduos. Para todos os sistemas foi ajustada a temperatura de anelamento para 50 ºC. Foi otimizada as condições da reação para cada sistema de multiplex. Na Figura 1 contém um painel com os sistemas multiplex, sendo 19 indivíduos de cada gel. O GenePop nos oferece uma inferência sobre o EHW, o déficit de heterozigotos e o excesso de heterozigotos através do teste de hipóteses e o número de alelos em cada locus. O Equilíbrio de Hardy-Weinberg consiste no equilíbrio de frequências genotípicas e alélicas. Tais valores estão na Tabela 2 para cada locus utilizados nos sistemas de multiplex. A população é considerada em equilíbrio quando possuem valor não- significativo. Com a correção de Holm-Bonferroni (α=0,0008) obtivemos valores mais precisos para ponderar se determinado locus está ou não em equilíbrio. O Equilíbrio de Hardy-Weinberg consiste na conformidade das frequências genotípicas observadas, com aquelas esperadas em uma população de acasalamentos aleatórios, dada uma frequência alélica. Os marcadores que não estão em EHW podem indicar alguns fenômenos populacionais como o efeito Wahlund onde a população é mista, contendo subestrutura. Metade dos marcadores da multiplex demonstraram EHW. Os resultados obtidos na Tabela 2 foram comparados aos descritos em Arias et al., 2016. Os marcadores Borb04, Borb09, Borb17, Borb24, Borb25, Borb33, Borb39, Borb44 estão em equilíbrio conforme a correção de Holm-Bonferroni com valores de p não-significativos. Os marcadores Borb01, Borb07, Borb08, Borb11, Borb15, Borb30, Borb36, Borb46 com valor de p significativo após a correção de Holm-Bonferroni não estão em equilíbrio. Essa comparação nos mostra que a utilização de sub-amostra de 36 indivíduos é suficiente para avaliação genética da população. Houve apenas uma diferença quando comparado a número de alelos no Borb07, o qual teve um alelo a menos na sub-amostra aqui analisada. Esses sistemas de multiplex são importantes para conservação e produção desse peixe, pois com a utilização destes ocorre uma diminuição de custos e uma maior celeridade nas análises genéticas, permitindo uma rápida obtenção de perfis moleculares de estoques naturais ou domesticados deste peixe.Conclusões
Foram desenvolvidos seis sistemas de multiplexes inéditos para B. orbignyanus; Os sistemas multiplex elaborados são capazes de acelerar em cerca de 2,5 vezes a análise destes 16 marcadores microssatélites; A análise dos seis multiplex teve performance comparável à análise dos 29 loci descritos em conjunto; e Estes resultados irão auxiliar na conservação e produção deste importante recurso pesqueiro.Gráficos e Tabelas