Genotipagem do gene kappa caseína e sua influência na porcentagem de caseína e produção de queijo frescal
2 - Universidade do Oeste Paulista
3 - Universidade do Oeste Paulista
4 - Universidade do Oeste Paulista
5 - Universidade do Oeste Paulista
RESUMO -
Os avanços tecnológicos da biologia molecular permitem genotipar o rebanho bovino de leite procurando desempenho melhor na maior produtividade e no rendimento dos teores de proteínas. O objetivo do presente estudo foi genotipar o gene K-CN em fêmeas das raças Holandesa, Girolando e Mestiços da fazenda experimental da Universidade do Oeste Paulista, por meio da técnica PCR- RFLP, e comparar o rendimento da produção de queijo frescal. Os resultados mostraram que o genótipo AB apresentou maior influência no rendimento da produção de queijo nas três raças estudadas (P=0,01). Com este estudo concluiu-se que as variações genéticas para K-CN podem ser usadas na aplicação da seleção assistida como marcadores buscando melhorar a produção de queijo frescal nos animais da raça Holandesa, Girolando e as vacas sem raça definida.
Genotyping of kappa casein gene and its influence on the percentage of casein and production of unripened cheese
ABSTRACT - The technological advances molecular biology allow to genotype bovine milk herd looking for better performance and yield of protein contents. The objective of the present study was genotype the K-CN gene in females breeds Dutch, Gir and Crossbreed of the experimental farm of Universidade do Oeste Paulista, through the PCR-RFLP technique, and compare yield fresh cheese production. The results showed that AB genotype had a greater influence on yield of cheese production the three studied races (P = 0.01). This study concluded that the genetic variations for K-CN can be used in the selection of assisted selection as markers to improve production the fresh cheese in the Dutch, Gir and crossbreed cows.Introdução
O Brasil é o quarto produtor mundial de leite de vaca, ficando atrás apenas de EUA, Índia e China. O leite está entre os produtos mais importantes da agropecuária nacional para economia, superado apenas por café e arroz. A qualidade e a produção do leite são influenciadas por diversos fatores como, os ambientais, a nutrição e fatores genéticos como raça e a fisiologia conforme a idade ao primeiro parto, período de lactação e ordem de parto (RIBEIRO et al., 2008).lAs proteínas presentes no leite são divididas em dois grandes grupos: as caseínas e as proteínas do soro. Há quatro tipos de caseínas: α1, α2, β e K. A caseína α1 compreende entre 39 a 46% do total das proteínas. A Kappa caseína constitui cerca de 25% da fração de caseína do leite e vários polimorfismos foram encontrados para esta proteína (PINTO et al., 2012).
A biologia molecular traz novas ferramentas nos processos de melhoramento animal o que, aliadas às metodologias tradicionais, contribuem para o progresso genético dos animais domésticos.
O objetivo deste estudo foi genotipar as fêmeas em idade reprodutiva do rebanho leiteiro da Universidade do Oeste Paulista - SP, através da técnica de PCR quanto ao gene K-CN e comparar os genótipos com os parâmetros físico-químicos do extrato seco e rendimento em caseína na produção de queijo frescal nas respectivas amostras de leite.
Revisão Bibliográfica
A produção de queijo é a principal atividade na indústria de laticínios, especialmente nas regiões Sul e Sudeste do Brasil, em decorrência disto é grande a preocupação em avaliar os principais indicadores de qualidade do leite e dos seus constituintes como, a gordura, proteína, lactose, extrato seco desengordurado, sólidos totais. Estas analises possuem o intuito de prevenir a qualidade final do leite e derivados oferecidos ao consumidor, e aumentar a competitividade do país no cenário de produção láctea (MOTTA et al., 2015). A Normativa 51 de 18/09/2002 classifica o leite cru produzido nos estabelecimentos leiteiros de acordo com o controle sanitário do rebanho, bem como padrões de higiene e a população microbiológica. Essa classificação ocorre em três categorias, Tipo A, Tipo B e Tipo C. Machado et al. (2002), Farrel et al. (2004) e Oliveira (2008) especificam a composição do leite em variadas substâncias como a como a lactose, sódio, magnésio e potássio, sais minerais, vitaminas, emulsão de gordura e suspensão coloidal de micelas de caseína ligada ao cálcio fósforo. As condições de coagulação influenciam diretamente na quantidade de minerais e proteínas presentes no queijo. As proteínas são um grupo de fosfoproteína dividida em quatro classes, das quais duas são sintetizadas na glândula mamária (β-lactoglobulina e a α-lactoalbumina), enquanto as outras são de origem sanguínea (albumina sérica e imunoglobulina), estas são constituídas de micelas formadas por submicelas, agregadas por interações hidrofóbicas e pontes salinas sofrendo precipitação em pH 4,6 (FARREL et al.,2004). A identificação de genes polimórficos que codificam as principais caseínas pode permitir melhorar a compreensão do comportamento do leite durante o processamento na indústria, sendo úteis como marcadores genéticos para critérios de seleção e cruzamentos de animais de gado leiteiro. Além disso, investigações de associações entre polimorfismo e características quantitativas e qualitativas do leite podem direcionar sua utilização pela indústria (STIPP et al., 2013).Materiais e Métodos
A presente pesquisa foi realizada na fazenda experimental da Universidade do Oeste Paulista em Presidente Prudente - SP, com 18 animais em idade reprodutiva sendo 6 vacas da raça Holandês, intituladas de grupo H, 6 vacas da raça Girolando referente ao grupo G e 6 vacas de raça Mestiça, que fazem parte do grupo S. Foram coletados 5 mL de sangue venoso de cada animal, em tubos a vácuo contendo EDTA. A identificação do DNA genômico nas amostras de sangue foi feita por meio da reação da cadeia polimerase em termociclador. Para o gene da K-CN, os “primers” utilizados foram o BKC1: 5’ ATCATTTATGGGCCATTCCACCAAAG 3’ e o BCK3: 5’ TTAGCCCATTTCGCTTCTCTGTAACA 3’ para a amplificação de um fragmento de 350pb. A solução de reação conteve 100ng de DNA, 10% de tampão para PCR (20 mM Tris-HCl, pH 8.4, 50mM KCl), 1,5mM MgCl2, 0,2mM de DNTP, 0,4mM de cada “primer” e 1 U de Taq DNA polimerase, totalizando 25mL. As reações para o gene da K-CN ocorreram em 44 ciclos e as temperaturas empregadas foram: desnaturação a 95ºC por 1 minuto; anelamento a 52ºC por 1 minuto; extensão a 72ºC por 1 minuto e 30 segundos; e extensão final a 72ºC por 4 minutos. O produto da amplificação sofreu digestão pela enzima Hinf I, nas seguintes condições: 10mL do produto do PCR, 1,25 U da enzima, 10% do tampão de reação e água mili-Q totalizando 13mL, que foi incubado por três horas a 37ºC, conforme método adaptado. Após a digestão enzimática, 13mL as amostras foram submetidas à corrida de eletroforese por duas horas em gel de agarose a 2% corado com 2mL de Brometo de Etídio (10mg/mL). Para a visualização das amostras, utilizamos o corante azul de Bromofenol (0,01%). O resultado foi observado com iluminação ultravioleta em câmara escura e as imagens capturadas por uma câmera Alpha-Innotech, utilizando-se o QUANTUM CAPT. Foi coletado também o volume de um litro de leite por animal em recipiente limpo de plástico e refrigerado a 4ºC, e as análises físico-química foram realizadas em forma de triplicata para cada litro colhido, totalizando 54 amostras. Para a determinação de caseína, foi utilizado o protocolo de ZENEBON (2008) com adaptações. O rendimento das fabricações dos queijos foi expresso em gramas de sólidos totais de queijo por litro de leite (g ST/l).Os resultados foram comparados por meio da Análise de Variância (ANOVA) e em caso de diferença significativa foi utilizado o teste de Tukey a 5% de significância (p<0,05). As análises estatísticas foram realizadas no programa BioEstat 5.0.Resultados e Discussão
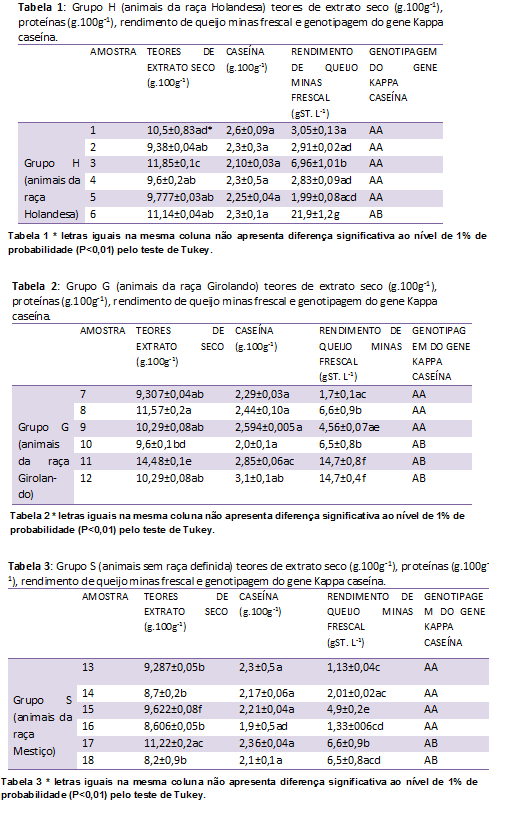
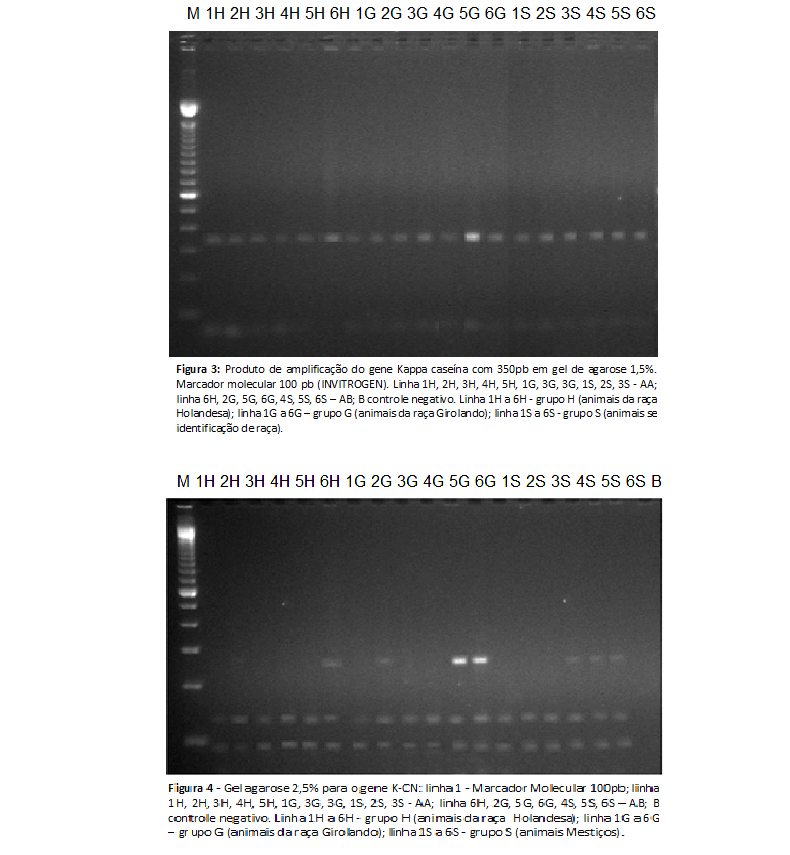
As frequências genotípicas foram estimadas por contagem dos genótipos visualizados na eletroforese obtida com o uso da técnica de PCR- RFLP e os padrões de migração se repetiram evidenciando a detecção do polimorfismo. Por meio da técnica de PCR foi possível a amplificação de um seguimento do gene K-CN com 350pb, correspondente aos “primers” desenhados e com resultados da literatura para o gene em questão (Figura 3). Após a amplificação do gene K-CN, foi realizado o tratamento de digestão com a enzima Hinf I, e identificado dois sítios de restrição no produto de amplificação do gene da Kappa Caseína para o alelo A; posição 134pb e outro na posição 266pb; no entanto o alelo B apenas no último sítio foi detectado. A digestão do alelo A produz bandas com 84pb e 132/134pb, e o alelo B resulta em fragmentos de restrição com 84pb e 266pb, o alelo AB resulta em fragmentos de restrição com 84pb, 132/134pb/ 266pb (figura 4). Os resultados demonstraram que 11 amostras apresentaram o genótipo AA, 7 apresentaram o genótipo AB e nenhuma apresentou o genótipo BB. De acordo com os resultados os teores de extrato seco não obtiveram diferença significativa entre os 6 animais do mesmo grupo. A média do grupo foi de 10,37%, a Tabela 2 mostra uma média de extrato seco de 10,92% e a Tabela 3 9,27%. A caseína foi o parâmetro seguinte a ser analisada, no grupo H a média entre os animais de genótipo AA tiveram 23% assim como o animal com genótipo AB. A média estimada é de 25% (ZENEBON et al., 2008). Os demais grupos apresentaram mesmo perfil entre as amostras. Em relação ao rendimento de queijo relacionado ao genótipo AB, a amostra 6 teve seis vezes mais rendimento em relação ao genótipo AA. No grupo G os animais com genótipos AB foram três vezes superiores em relação aos genotipados AA. O grupo S demonstrou que os animais AB foram três vezes mais produtivos em termos de rendimento de queijo quando comparados aos animais AA. De modo geral o grupo H apresentou maior rendimento do queijo frescal com 25,1%. O grupo S foi o que menos teve rendimento com apenas 8,74%. O genótipo BB está associado a características de processamento superior para produção de queijo, com menor tempo de coagulação e formação de coágulo com maior densidade, resultando assim em maior produção. Em relação aos animais BB, apresentam rendimento 12% superior de caseína encontrada no fabrico do queijo muçarela e 8% no fabrico do queijo tipo cheddar em relação aos animais com o genótipo AA. Os animais AB apresentam rendimento intermediário entre os genótipos BB e AA. Animais AA possuem o genótipo menos favorável para produção de queijo. STIPP, (2013) ao analisar a frequência genotípica AA, AB e BB, por meio das mesmas técnicas descritas neste trabalho, associando com a produção de leite (kg leite/dia) em bovinos das raças Girolanda, Holandesa e Jersey, concluiu que o alelo B da K-CN apresentou maior produtividade leiteira nas raças Girolanda e Holandesa. A alta periodicidade do alelo A do gene da k-CN na raça Girolanda possivelmente está relacionada à sua origem em cruzamentos das raças Gir e Holandesa. Deb (2014) pesquisou o polimorfismo da kappa caseína em 200 animais e observou que o alelo A foi mais frequente do que o alelo B entre a população de Frieswal.Os referidos autores notaram que os animais com alelos AB tiveram um maior rendimento de leite total no período de 300 dias quando comparados aos do genótipo AA. Oner & Elmaci, (2009) relacionou um maior rendimento de queijos nos genótipos k-caseína (BB), β-caseína (A²A²) e β-lactoglobulina (AA)..Dentre os animais avaliados não foi possível detectar o alelo homozigoto BB, ao contrário do encontrado por Ng-Kwai-Hang, (2004) que observaram superior produção leiteira para as vacas holandesas com o genótipo BB.Conclusões
De acordo com as análises realizadas, foi possível verificar associação do alelo B com a maior produção de Queijo minas frescal nos três grupos avaliados. Não ocorreram mudanças significativas quanto aos valores de caseína e de teores de extrato seco quando relacionado com o genótipo identificado. O conhecimento desses dados genotípicos concede um direcionamento mais adequado dos cruzamentos e a seleção prematura de animais, auxiliando substancialmente para a melhoria na produção de queijo minas frescal.Gráficos e Tabelas