POLIMORFISMO DO GENE MITOCONDRIAL CITOCROMO C OXIDASE SUBUNIDADE 1 (COI) NA ESPÉCIE Loricariichthys anus.
Mateus Tremea1, Suellen Susin Gazzola2, Suele Bueno dos Santos3, Vanessa Seidel4, Alexandra Moller Alves5, Gadriéli Cristina Gheno6, Rafael Aldrighi Tavares7, Sergio renato Noguez Piedras8
1 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
2 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
3 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
4 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
5 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
6 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
7 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
8 - Universidade Federal de Pelotas UFPEL
2 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
3 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
4 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
5 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
6 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
7 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
8 - Universidade Federal de Pelotas UFPEL
RESUMO -
Objetivou-se identificar o polimorfismo da região mitocondrial Citocromo Oxidase Subunidade I (COI) da espécie Loricariichthys anus. A montagem por referência foi realizado com o programa Bowtie2, sendo utilizado como referência o gene COI da espécie Loricariichthys anus. Os totais de 121 reads alinharam a 652 pares de base do gene de referência, revelando 13 sítios de polimorfismo. A partir do sequenciamento de nova geração identificou-se o polimorfismo do gene COI da espécie Loricariichthys anus, sendo de grande relevância para base de trabalhos futuros de conservação e melhoramento genético.
Palavras-chave: polimorfismo, COI, mtDNA.
POLYMORPHISM OF OXIDASE CYTOCHROME C SUBUNIT I MITOCHONDRIAL GENE (COI) ON Loricariichthys anus.
ABSTRACT - The objective of the study was to identify the polymorphism of Oxidase Cytochrome Subunit I mitochondrial region (COI) on Loricariichthys anus. The reference assembly was done with Bowtie2 program, using the COI gene of Loricariichthys anus as reference. The amount of 121 reads was aligned to 652 base pairs of the reference gene, revealing 13 polymorphism sites. From the sequencing of a new generation, the polymorphism on the COI gene was identified, having great relevance as a basis for future works on conservation and genetic improvement.Keywords: polymorphism, COI, mtDNA.
Introdução
A espécie Viola da Lagoa, Loricariichthys anus, é uma das espécies mais numerosas nas Lagoas da região Sul do Brasil, apresenta grandes perspectivas à aquicultura pelo alto valor de sua carne além de ser uma espécie muito utilizada como alimento pelas populações ribeirinhas de diversas regiões do Rio Grande do Sul. A molécula do DNA mitocondrial (mtDNA) passou a fazer parte de muitos, senão da maioria dos estudos envolvendo estrutura populacional, relações filogenéticas e o entendimento de vários aspectos biológicos e evolutivos de uma grande variedade de organismos desde as décadas de 70-80, do século passado (WILSON et al. 1985; AVISE et al. 1987; MORITZ et al. 1987; AVISE 1993). O seguinte trabalho teve como objetivo identificar o polimorfismo do gene mitocondrial citocromo oxidase subunidade 1 (COI) na espécie Loricariichthys anus, indicando a variabilidade existente no indivíduo e na espécie.Revisão Bibliográfica
A viola Loricariichthys anus é uma das principais espécies capturadas por pescadores artesanais da Lagoa Mangueira. Pertence à família Loricariidae, e segundo Querol (1996) apresenta grandes perspectivas para a aquicultura pelo alto valor de sua carne e pela adaptação a ambientes lênticos com pouca exigência quanto ao teor de oxigênio. O gene mitocondrial COI demonstra ser um importante marcador molecular para código de barras genético de animais, pois oferece a vantagem de ser espécie-específico (Hebert, Ratnasingham & Waard, 2003), i.e. apresenta alta variabilidade interespecífica. Essa característica tornou o COI, desde então, um marcador universal em taxonomia molecular e isso inspirou a criação de um consórcio internacional conhecido como Código de Barras da Vida (do inglês, Barcode of Life – http://www.barcodeoflife.org). Esse projeto é mantido em parceria com três importantes bases de dados moleculares: GenBank (http://www. ncbi.nlm.nih.gov), European Molecular Biology Lab (http://www.ebi.ac.uk/embl) e DNA Data Bank of Japan (http://www.ddbj.nig.ac.jp). A utilização de um gene mitocondrial, Citocromo Oxidase Subunidade I (COI), que faz parte do complexo gênico codante de proteínas transmembranares, envolvidas no transporte elétrico e catálise da cadeia respiratória de organismos eucariotos, está sendo utilizado em estudos de população, variabilidade genética e filogenia.Materiais e Métodos
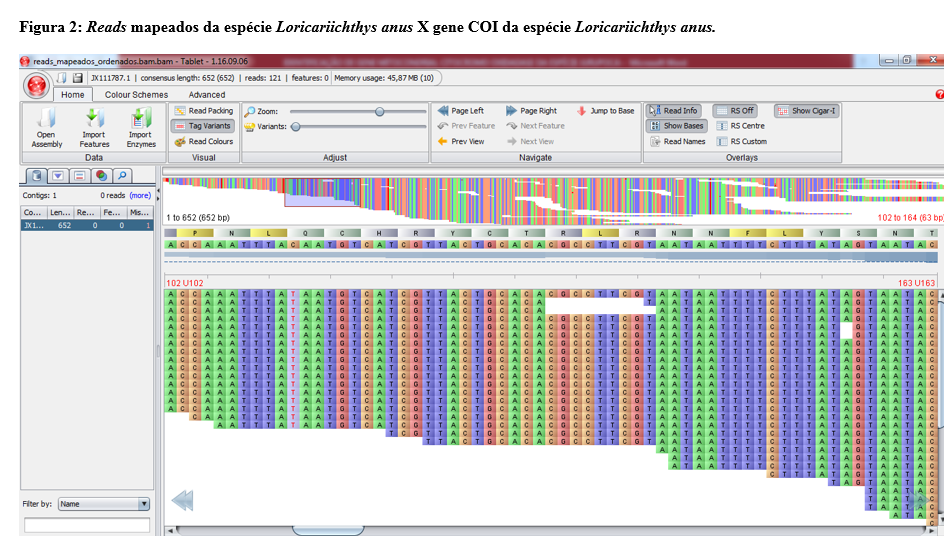
A biblioteca de DNA, da espécie Loricariichthys anus foi obtida a partir de um sequenciador GAIIx (Illumina, USA) no modo paired-end, para a obtenção de sequências com 150 pares de bases (pb). A qualidade de cada read foi analisada com o programa FastQC (PATEL e JAIN, 2012). Após a análise de qualidade foi realizada a remoção dos adaptadores e a remoção de read de baixa qualidade com o programa Trimmomatic (BOLGER et al., 2014). As sequências das extremidades dos reads foram removidas quando as médias de qualidade fossem inferiores a Phread 15 em intervalos de quatro bases. Também foram removidos os reads com comprimentos menores que 32pb. Para a verificação da eficiência da filtragem foi utilizado novamente o programa FastQC. A montagem por referência foi realizado com o programa Bowtie2 (LANGMEAD e SALZBERG, 2012) sendo utilizado como referência o gene mitocondrial COI da espécie Loricariichthys anus (GenBank: JX111787.1) com comprimento de 652pb. O arquivo no formato SAM, gerado na montagem, foi transformado em formato BAM com o programa Samtoll (LI et al., 2009) e posteriormente as reads montadas contra a referência foram visualizadas com o programa Tablet (MILNE et al., 2013).Resultados e Discussão
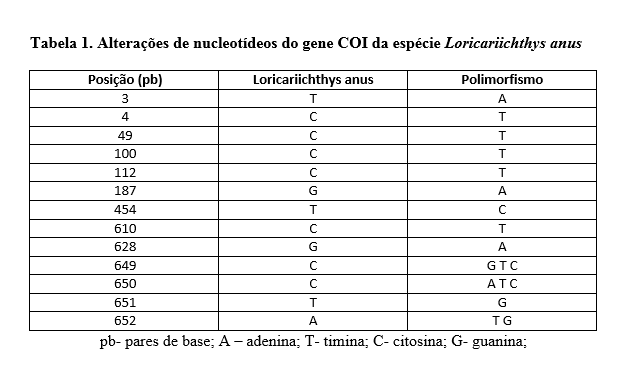
O sequenciamento do DNA resultou em aproximadamente 1.103.580 reads, sendo estes de 35 a 151pb, subdivididos em dois arquivos paired-end. Subsequentemente fez-se a filtragem através do programa Trimmomatic mantendo-se 1.059.512 reads e sendo removidos 44.068 reads. As bases que restaram apresentam-se de boa qualidade, com valores de Phred acima de 24. Segundo DEL FABBRO et al. (2013), ocorre o aumento das taxas de alinhamento dos reads a um genoma de referência, mesmo com a diminuição do volume de sequências após a filtragem. Os totais de 121 reads alinharam a 652 pares de base do gene de referência COI de Loricariichthys anus (figura 2), mostrando 13 sítios de polimorfismo (tabela 1). O COI é o marcador molecular mais utilizado, devido a sua organização genômica simples e uniforme, à falta de recombinação e de taxas elevadas de substituições nucleotídicas, além da possibilidade de recuperação eficaz de informações gênicas de amostras em estados inadequados de preservação (ZHANG & HEWITT, 1997).Conclusões
A partir do sequenciamento de nova geração identificou-se o polimorfismo do gene mitocondrial citocromo oxidase subunidade 1 (COI) da espécie Loricariichthys anus. Caracterizar regiões mitocondriais é de grande importância para base de trabalhos futuros de conservação e melhoramento genético.Gráficos e Tabelas