POLIMORFISMO DO GENE MITOCONDRIAL CITOCROMO OXIDASE SUBUNIDADE I DA ESPÉCIE JURUPOCA (Hemisorubim phatyrhynchos).
Mateus Tremea1, Suellen Susin Gazzola2, Suele Bueno dos Santos3, Gadriéli Cristina Gheno4, Alexandra Moller Alves5, Vanessa Seidel6, Rafael Aldrighi Tavares7, Heden Luiz Marques Moreira8
1 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
2 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
3 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
4 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
5 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
6 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
7 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
8 - Universidade Federal de Pelotas UFPEL
2 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
3 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
4 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
5 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
6 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
7 - Universidade Federal de Santa Maria UFSM, campus Palmeira das Missões
8 - Universidade Federal de Pelotas UFPEL
RESUMO -
Objetivou-se identificar o polimorfismo da região mitocondrial Citocromo Oxidase Subunidade I (COI) da espécie Hemisorubim phatyrhynchos. O programa Bowtie2 foi utilizado para realizar a montagem de referência, o gene mitocondrial utilizado como referência foi o da espécie Hemisorubim phatyrhynchos. Um total de 333 reads foram alinhados a 685 pares de base do gene de referência, revelando 31 sítios de diferenciação entre as espécies. A partir do sequenciamento de nova geração foi identificado o polimorfismo do gene mitocondrial Citocromo Oxidase Subunidade I (COI) da espécie Hemisorubim phatyrhynchos, sendo de grande relevância para base de trabalhos futuros de conservação e melhoramento genético.
Palavras-chave: Citocromo Oxidase Subunidade I (COI); DNA mitocondrial; Jurupoca.
POLYMORPHISM OF OXIDASE CYTOCHROME SUBUNIT I MITOCHONDRIAL GENE ON JURUPOCA SPECIES (Hemisorubim phatyrhynchos).
ABSTRACT - The objective of the study was to identify the polymorphism of Oxidase Cytochrome Subunit I mitochondrial region (COI) of Hemisorubim phatyrhynchos. The Bowtie2 program was applied to do the reference assembly and the mitochondrial gene used as reference was the Hemisorubim phatyrhynchos. A number of 333 reads was aligned to 685 base pairs of the reference gene, revealing 31 differentiation sites between the species. From the sequencing of a new generation, the polymorphism of Oxidase Cytochrome Subunit I mitochondrial gene (COI) on Hemisorubim phatyrhynchos was identified, having great relevance as a basis for future works on conservation and genetic improvement.Keywords: Oxidase Cytochrome Subunity I (COI), mitochondrial DNA, jurupoca.
Introdução
Hemisorubim platyrhynchos, popularmente conhecida como jurupoca, é uma espécie de médio porte, pertencente à família Pimelodidae e tem ocorrência nos grandes rios da América do Sul, nas bacias dos rios Amazonas, Maroni, Orinoco e Paraná (REIS et al., 2003). O genoma mitocondrial dos animais é, na grande maioria dos casos, constituído por uma molécula de DNA circular, pequena, com conteúdo gênico conservado (apenas 37 genes) e estrutura gênica simples (não possui DNA repetitivo) (Wilson et al., 1985). Essa extrema economia de conteúdo da maioria dos genomas mitocondriais animais é atribuída a uma intensa seleção a favor de um genoma pequeno (Moritz et al., 1987). Por outro lado, essa molécula apresenta alta taxa de evolução por mutação, cerca de 5 a 10 vezes mais rápida do que a taxa de mutação de um gene nuclear de cópia única (Moritz et al., 1987). A combinação dessas características faz com que o DNAmt seja amplamente utilizado em estudos de caracterização de populações, subespécies e espécies, além de estudos de caráter evolutivo e filogenético(Harrison, 1989). Com este trabalho objetivou-se identificar o polimorfismo do gene mitocondrial COI da espécie Hemisorubim platyrhynchos, relevando a variabilidade existente no indivíduo, e consequentemente, na espécie.Revisão Bibliográfica
A espécie jurupoca é predadora de microfauna bentônica e de peixes, com uma longevidade de 11,4 anos, podendo alcançar até 64 cm de comprimento (PENHA et al., 2003). Trata-se de uma espécie carnívora que alimenta-se de peixes e invertebrados, sua carne é de tonalidade amarelada de excelente sabor, sendo muito apreciada. O gene mitocondrial COI demonstra ser um importante marcador molecular para código de barras genético de animais, pois oferece a vantagem de ser espécie-específico (Hebert, Ratnasingham & Waard, 2003), i.e. apresenta alta variabilidade interespecífica. Essa característica tornou o COI, desde então, um marcador universal em taxonomia molecular e isso inspirou a criação de um consórcio internacional conhecido como Código de Barras da Vida (do inglês, Barcode of Life – http://www.barcodeoflife.org). Esse projeto é mantido em parceria com três importantes bases de dados moleculares: GenBank (http://www. ncbi.nlm.nih.gov), European Molecular Biology Lab (http://www.ebi.ac.uk/embl) e DNA Data Bank of Japan (http://www.ddbj.nig.ac.jp). Há indícios de que o gene mitocondrial Citocromo C Oxidase I (COI) tem força para aumentar significativamente o número de espécies catalogadas, além disso, ele pode ainda resolver até 95% dos casos de espécies em avaliação (Armstrong e Ball, 2005). Esse motivo se dá porque o gene COI apresenta pequena variação intraespecífica (Herbert e Gregory, 2005).Materiais e Métodos
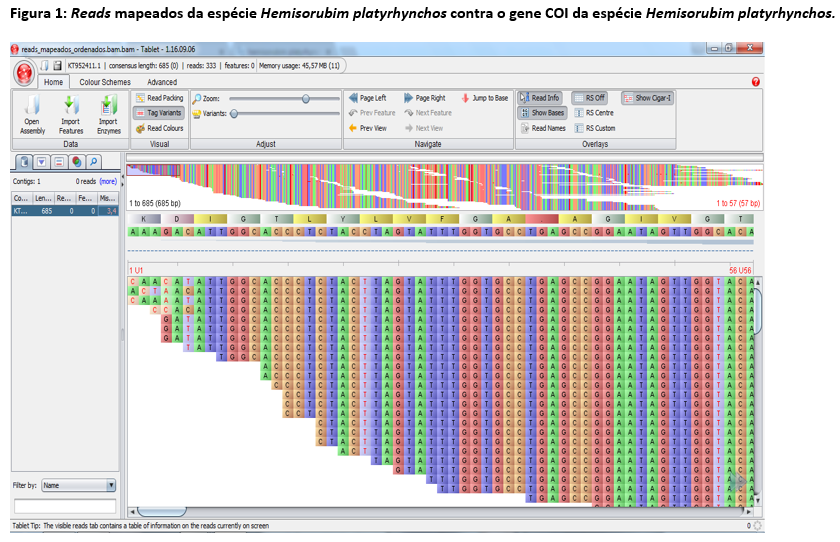
A biblioteca de DNA, da espécie Hemisorubim platyrhynchos foi obtida a partir de um sequenciador GAIIx (Illumina, USA) no modo paired-end, para a obtenção de sequências com 150 pares de bases (pb). A qualidade de cada read foi analisada com o programa FastQC (PATEL e JAIN, 2012). Após a análise de qualidade foi realizada a remoção dos adaptadores e a remoção de read de baixa qualidade com o programa Trimmomatic (BOLGER et al., 2014). As sequências das extremidades dos reads foram removidas quando as médias de qualidade fossem inferiores a Phread 15 em intervalos de quatro bases. Também foram removidos os reads com comprimentos menores que 32pb. Para a verificação da eficiência da filtragem foi utilizado novamente o programa FastQC. A montagem por referência foi realizado com o programa Bowtie2 (Langmead e Salzberg, 2012) sendo utilizado como referência o gene mitocondrial Citocromo oxidase I (GenBank: KT952411.1) com comprimento de 685 pb da espécie Hemisorubim platyrhynchos. O arquivo no formato SAM, gerado na montagem, foi transformado em formato BAM com o programa Samtoll (LI et al., 2009) e posteriormente a reads montadas contra a referência foram visualizadas com o programa Tablet (MILNE et al., 2013).Resultados e Discussão
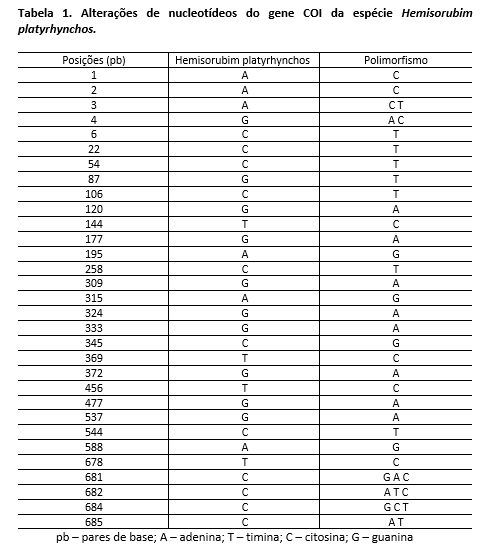
O sequenciamento de DNA resultou em aproximadamente 1.816.200 reads sendo esses, reads, sendo estes de 35 a 151pb, subdivididos em dois arquivos paired-end. Após foi realizada a filtragem através do programa Trimmomatic mantendo-se 1.762.764 reads e sendo removidos 53.436 reads. As bases que restaram apresentam-se de boa qualidade, com valores de Phred acima de 26. Segundo Del Fabbro et al. (2013), ocorre o aumento das taxas de alinhamento dos reads a um genoma de referência, mesmo com a diminuição do volume de sequências após a filtragem. Houve um total de 333 reads alinhados aos 685 pares de base do gene de referência COI da espécie Hemisorubim platyrhynchos (figura 1), Ocorreram 31 sítios de polimorfismo (tabela 1). A utilização de um gene mitocondrial, Citocromo Oxidase Subunidade I (COI), que faz parte do complexo gênico codante de proteínas transmembranares, envolvidas no transporte elétrico e catálise da cadeia respiratória de organismos eucariotos, está sendo utilizado em estudos de população, variabilidade genética e filogenia.Conclusões
A partir do sequenciamento de nova geração identificou-se o polimorfismo do gene mitocondrial Citocromo Oxidase Subunidade I (COI) da espécie Hemisorubim platyrhynchos. É de grande relevância caracterizar regiões mitocondriais para base de trabalhos futuros de conservação e melhoramento genético.Gráficos e Tabelas